나선형 나선형
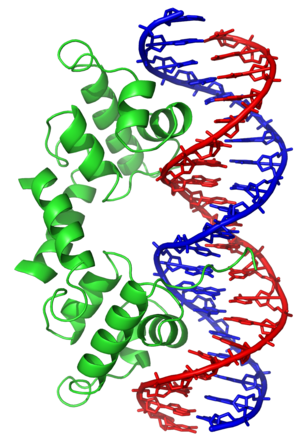
Helix-turn-helixHelix-turn-helix는 DNA결합단백질(DBP)이다.HTH(Helix-Turn-Helix)는 DNA와 결합할 수 있는 주요 구조적 모티브이다.각 단량체는 DNA의 주요 홈에 결합하는 짧은 아미노산 가닥에 의해 결합된 두 개의 α 나선형 구조를 포함한다.HTH 모티브는 유전자 발현을 조절하는 많은 단백질에서 발생한다.나선-루프-나선 [1]모티브와 혼동해서는 안 된다.
검출
나선-돌기-나선 모티브의 발견은 박테리오파지 람다의 전사 조절 단백질을 코드하는 여러 유전자(Cro, CAP 및 γ 억제제) 사이의 유사성에 기초했으며, 이들은 DNA [2][3][4][5]인식을 용이하게 하는 공통 20-25개의 아미노산 배열을 공유하는 것으로 밝혀졌다.
기능.
나선-회전-나선 모티브는 DNA 결합 모티브입니다.나선-돌기-나선 단백질에 의한 DNA 인식 및 결합은 모티브의 N 말단을 차지하고 다른 하나는 C 말단에서 차지하는 두 개의 α 나선에 의해 이루어진다.크로 억제제와 같은 대부분의 경우, 두 번째 나선은 DNA 인식에 가장 많이 기여하기 때문에 종종 "인식 나선"이라고 불린다.그것은 일련의 수소 결합과 노출된 염기와의 다양한 반데르발스 상호작용을 통해 DNA의 주요 홈에 결합합니다.다른 α나선은 단백질과 DNA 사이의 상호작용을 안정시키지만,[2] 그 인식에는 특별히 강한 역할을 하지 않는다.인식 나선과 그 앞의 나선은 항상 같은 상대 [6]방향을 가진다.
나선-회전-나선 모티브 분류
나선형 [6][7][8]모티브의 구조와 나선형의 공간 배열을 기반으로 분류하려는 여러 시도가 있었다.주요 유형 중 일부를 아래에 설명합니다.
디헬리컬
이나선모티브는 가장 단순한 나선모티브다선모티브는 나선모티브다.두 개의 나선형만을 포함하는 Engraded 호메오도메인의 단편은 초고속 독립적으로 접히는 단백질 [9]도메인으로 밝혀졌다.
세 개의 나선형
이 모티브의 예는 전사활성제 Myb에서 [10]찾을 수 있다.
사나선
테트라 나선 나선-턴-나선 모티브는 3 나선 모티브와 비교하여 C 말단 나선을 추가로 가진다.여기에는 세균전사인자에서 발견되는 LuxR형 DNA결합 HTH 도메인과 TetR [11]억제제에서 발견되는 나선-턴-나선 모티브가 포함된다.추가 나선형이 있는 다중 나선형 버전도 발생합니다.[12]
날개나선
날개 달린 나선-턴-나선(wHTH) 모티브는 3 나선 다발과 3 또는 4 스트랜드 베타 시트(날개)로 형성된다.wHTH 모티브에서 나선 및 가닥의 토폴로지는 다를 수 있습니다.ETS wHTH는 제3나선이 DNA 인식나선인 [13][14]α1-β1-β2-α2-β3-β4 순서로 배열된 4가닥 반평행 베타시트 비계상에서 나선회전나선 모티브로 접힌다.
기타 수정된 나선-회전-나선 모티브
나선-턴-나선 모티브의 다른 유도체로는 다수의 항생제 내성의 조절제인 MarR에서 발견된 DNA 결합 도메인이 있으며, 이는 추가적인 C 말단 알파 나선을 [8][15]가진 날개 달린 나선-턴-나선을 형성한다.
「 」를 참조해 주세요.
레퍼런스
- ^ Brennan RG, Matthews BW (February 1989). "The helix-turn-helix DNA binding motif". The Journal of Biological Chemistry. 264 (4): 1903–6. doi:10.1016/S0021-9258(18)94115-3. PMID 2644244.
- ^ a b Matthews BW, Ohlendorf DH, Anderson WF, Takeda Y (March 1982). "Structure of the DNA-binding region of lac repressor inferred from its homology with cro repressor". Proceedings of the National Academy of Sciences of the United States of America. 79 (5): 1428–32. Bibcode:1982PNAS...79.1428M. doi:10.1073/pnas.79.5.1428. PMC 345986. PMID 6951187.
- ^ Anderson WF, Ohlendorf DH, Takeda Y, Matthews BW (April 1981). "Structure of the cro repressor from bacteriophage lambda and its interaction with DNA". Nature. 290 (5809): 754–8. Bibcode:1981Natur.290..754A. doi:10.1038/290754a0. PMID 6452580. S2CID 4360799.
- ^ McKay DB, Steitz TA (April 1981). "Structure of catabolite gene activator protein at 2.9 A resolution suggests binding to left-handed B-DNA". Nature. 290 (5809): 744–9. doi:10.1038/290744a0. PMID 6261152. S2CID 568056.
- ^ Pabo CO, Lewis M (July 1982). "The operator-binding domain of lambda repressor: structure and DNA recognition". Nature. 298 (5873): 443–7. Bibcode:1982Natur.298..443P. doi:10.1038/298443a0. PMID 7088190. S2CID 39169630.
- ^ a b Wintjens R, Rooman M (September 1996). "Structural classification of HTH DNA-binding domains and protein-DNA interaction modes". Journal of Molecular Biology. 262 (2): 294–313. doi:10.1006/jmbi.1996.0514. PMID 8831795.
- ^ Suzuki M, Brenner SE (September 1995). "Classification of multi-helical DNA-binding domains and application to predict the DBD structures of sigma factor, LysR, OmpR/PhoB, CENP-B, Rapl, and Xy1S/Ada/AraC". FEBS Letters. 372 (2–3): 215–21. doi:10.1016/0014-5793(95)00988-L. PMID 7556672. S2CID 3037519.
- ^ a b Aravind L, Anantharaman V, Balaji S, Babu MM, Iyer LM (April 2005). "The many faces of the helix-turn-helix domain: transcription regulation and beyond". FEMS Microbiology Reviews. 29 (2): 231–62. doi:10.1016/j.femsre.2004.12.008. PMID 15808743.
- ^ Religa TL, Johnson CM, Vu DM, Brewer SH, Dyer RB, Fersht AR (May 2007). "The helix-turn-helix motif as an ultrafast independently folding domain: the pathway of folding of Engrailed homeodomain". Proceedings of the National Academy of Sciences of the United States of America. 104 (22): 9272–7. Bibcode:2007PNAS..104.9272R. doi:10.1073/pnas.0703434104. PMC 1890484. PMID 17517666.
- ^ Ogata K, Hojo H, Aimoto S, Nakai T, Nakamura H, Sarai A, Ishii S, Nishimura Y (July 1992). "Solution structure of a DNA-binding unit of Myb: a helix-turn-helix-related motif with conserved tryptophans forming a hydrophobic core". Proceedings of the National Academy of Sciences of the United States of America. 89 (14): 6428–32. Bibcode:1992PNAS...89.6428O. doi:10.1073/pnas.89.14.6428. PMC 49514. PMID 1631139.
- ^ Hinrichs W, Kisker C, Düvel M, Müller A, Tovar K, Hillen W, Saenger W (April 1994). "Structure of the Tet repressor-tetracycline complex and regulation of antibiotic resistance". Science. 264 (5157): 418–20. Bibcode:1994Sci...264..418H. doi:10.1126/science.8153629. PMID 8153629.
- ^ Iwahara J, Clubb RT (November 1999). "Solution structure of the DNA binding domain from Dead ringer, a sequence-specific AT-rich interaction domain (ARID)". The EMBO Journal. 18 (21): 6084–94. doi:10.1093/emboj/18.21.6084. PMC 1171673. PMID 10545119.
- ^ Donaldson LW, Petersen JM, Graves BJ, McIntosh LP (January 1996). "Solution structure of the ETS domain from murine Ets-1: a winged helix-turn-helix DNA binding motif". The EMBO Journal. 15 (1): 125–34. doi:10.2210/pdb1etc/pdb. PMC 449924. PMID 8598195.
- ^ Sharrocks AD, Brown AL, Ling Y, Yates PR (December 1997). "The ETS-domain transcription factor family". The International Journal of Biochemistry & Cell Biology. 29 (12): 1371–87. doi:10.1016/S1357-2725(97)00086-1. PMID 9570133.
- ^ Alekshun MN, Levy SB, Mealy TR, Seaton BA, Head JF (August 2001). "The crystal structure of MarR, a regulator of multiple antibiotic resistance, at 2.3 A resolution". Nature Structural Biology. 8 (8): 710–4. doi:10.1038/90429. PMID 11473263. S2CID 19608515.
추가 정보
- Struhl K (April 1989). "Helix-turn-helix, zinc-finger, and leucine-zipper motifs for eukaryotic transcriptional regulatory proteins". Trends in Biochemical Sciences. 14 (4): 137–40. doi:10.1016/0968-0004(89)90145-X. PMID 2499084.
- Gajiwala KS, Burley SK (February 2000). "Winged helix proteins". Current Opinion in Structural Biology. 10 (1): 110–6. doi:10.1016/S0959-440X(99)00057-3. PMID 10679470.
- Santos CL, Tavares F, Thioulouse J, Normand P (March 2009). "A phylogenomic analysis of bacterial helix-turn-helix transcription factors". FEMS Microbiology Reviews. 33 (2): 411–29. doi:10.1111/j.1574-6976.2008.00154.x. PMID 19076237.
- Hoskisson PA, Rigali S (2009). "Chapter 1: Variation in form and function the helix-turn-helix regulators of the GntR superfamily". Advances in Applied Microbiology. 69: 1–22. doi:10.1016/S0065-2164(09)69001-8. PMID 19729089.
- Brennan RG (September 1993). "The winged-helix DNA-binding motif: another helix-turn-helix takeoff". Cell. 74 (5): 773–6. doi:10.1016/0092-8674(93)90456-Z. PMID 8374950. S2CID 31355349.
- Huffman JL, Brennan RG (February 2002). "Prokaryotic transcription regulators: more than just the helix-turn-helix motif". Current Opinion in Structural Biology. 12 (1): 98–106. doi:10.1016/s0959-440x(02)00295-6. PMID 11839496.
외부 링크
- EMBL의 Helix-Turn-Helix 모티브, 람다와 같은 억제기
- PDB ID 1LMB의 전체 PDB 엔트리
- Cro/C1 타입의 HTH 도메인, PROSITE의 HTH 증가