Bjelančevine


Ovo je prvi protein čija je struktura razjašnjena putem X-kristalografije.
Desno od centra među namotajima je proteinska hem grupa (prikazano u sivoj boji) s molekulom vezanog kisika (crvena).
Bjelančevine ili proteini su makromolekule (macro=mnogo, više; moliculis=sićušan, mali;) koje su nastale međusobnim spajanjem aminokiselina. Dogovoreno je da se spojevi koji broje manje od 50 aminokiselina u niz nazivaju polipeptidi, a da se makromolekule sa preko 50 aminokiselina nazivaju proteini. Bjelančevine su vrlo važni sastavni dijelovi svakog organizma, jer čine neke strukture i supstance koje su neophodne za život. Bjelančevine se dijele na proste (proteine) i složene bjelančevine (proteide). Složene bjelančevine (proteidi), osim proteinskog dijela imaju i prostetičku grupu. Prema hemijskom sastavu prostetičke grupe izvršena je podjela složenih bjelančevina na : nukleoproteide, hromoproteide, glikoproteide, fosfoproteide i lipoproteide.[1][2]
Pregled
[uredi | uredi izvor]Sinteza bjelančevina se dešava u ćelijama živih organizama, na specifičnim tjelašcima u ćeliji nazvanim ribosomi. Svaki protein ima svoj tačan niz aminokiselina, a informacija o njegovoj sintezi i osobenostima se nalazi unutar DNK organizma koji se posmatra. U DNK se nalazi specifičan kod koji definiše raspored i broj aminokiselina koje čine jedan protein. Promjenom redoslijeda samo jedne karike u lancu nastaće nova bjelančevina, potpuno novih osobina.[3]
Dakle, za neku specifičnu vrstu živog bića, osnovno je odabrati redosljed karika (aminokiselina) u lancu bjelančevina, kao i pravu smjesu za tu vrstu specifičnih bjelančevina. Tu zadaću ima DNK. Proteini, pored aminokiselina, mogu sadržavati i neke druge organske i neorganske materije npr. šećere – glikoproiteini, fosforne derivate – fosfoproteini, masti-lipoproteini. Svaka aminokiselina ima svoj amino i karboksilni dio, pa u spoju dvije ili više aminokiselina vežu se amino (-NH2) grupa jedne sa karboksilnom -(COOH) grupom druge aminokiseline i time nastaje hemijska veza nazvana peptidna veza -CO-NH-.
Sinteza proteina se odvija u kontinuiranom procesu koji ima nekoliko faza.
- Inicijacija je početna faza sinteze proteina, u kojoj počinje transkripcija genetičke informacije, uz katalitičko posredovanje odgovarajućih enzima. Tada enzim specifični antikodon iRNK prepoznaje komplementarni kodon na informacijskoj RNK i ugrađuje prvu aminokiselinu u buduči polipeptidni lanac.
- Elongacija je faza u kojoj se produžava lanac ugrađenih aminokiselina, zahvaljujući očitavanjem narednih tripleta u iRNK. Elongacija traje sve dotle dok se ne realizira kompletni redoslijed aminokiselina koje su kodirane genetičkom informacijom specifične iRNk, kao komplementarnom kopijom jednog polulanca odgovarajućeg lokusa.
- Terminacija molekule proteina nastaje nakon što iRNK ne realizira kodiranje posljednje aminokiseline u specifičnom polipeptidnom lancu (proteinu).
Struktura
[uredi | uredi izvor]Struktura bjelančevina je veoma složena i podijeljena je na nekoliko nivoa: primarna, sekundarna, tercijarna i kvaternerna struktura.
- Primarna struktura proteina podrazumijeva redoslijed vezanja aminokiselina u peptidnom lancu.
- Sekundarna struktura predstavlja izgled proteinskog lanca u prostoru (npr. alfa heliks). Za nju je odgovorna vodikova veza.
- Kod mnogih proteina dolazi do interakcija raznih funkcionalnih grupa u ostacima aminokiselina. To vodi do daljeg uvijanja, savijanja i zbijanja lanaca i takva trodimenzionalna struktura se zove tercijarna struktura. Zavisno od tercijarne strukture, proteini se dijele na fibrilarne i globularne.
- Fibrilarni proteini imaju vlaknastu strukturu i teško se otapaju u vodi.
- Globularni proteini imaju zbijenu strukturu loptastog oblika. Otapaju se u vodi i, zbog veličine molekula, formiraju koloide.
- Udruživanjem više proteina u veće agregate nastaje proteinski kompleks koji predstavlja kvaternernu strukturu.
Historija i etimologija
[uredi | uredi izvor]Kao posebnu klasu bioloških molekula, proteine su prepoznali Antoine Fourcroy i drugi, u osamnaestom stoljeću, koji su ih razlikovali po sposobnosti molekula da koaguliraju ili flokuliraju pod tretmanima toplotom ili kiselinom.[4] Najpoznatiji primjeri toga vremena uključivali su albumin iz bjelanjka, krvni serumski albumin, fibrin i pšenični gluten.
Proteine je prvi opisao holandski hemičar Gerardus Johannes Mulder, a imenovao ih je švedski hemičar Jöns Jacob Berzelius 1838.[5][6] Mulder je izvršio elementarnu analizu uobičajenih proteina i otkrio da skoro svi proteini imaju istu empirijsku formulu,C400H620N100O120P1S1.[7] Došao je do pogrešnog zaključka da bi oni mogli biti sastavljeni od jednog tipa (veoma velikih) molekula. Termin protein za opis ovih molekula predložio je Mulderov saradnik Berzelius; izveden je iz grčke riječi πρώτειος – proteios, što znači primarni,[8] "na čelu" ili "stoji ispred",[9] + sufiks-in. Mulder je nastavio da identifikuje proizvode razgradnje proteina kao što je aminokiselina leucin za koji je pronašao (skoro tačnu) molekulsku težinu od 131 Da.[7] Prije termina "protein", korišteni su drugi nazivi, poput "albumini" ili "albuminski materijali" ("Eiweisskörper", na njemačkom).[10]
Rani nutricionisti kao što je njemački Carl von Voit vjerovali su da je protein najvažniji nutrijent za održavanje strukture tijela, jer se općenito vjerovalo da "meso čini meso".[11] Karl Heinrich Ritthausen proširio je poznate oblike proteina identifikacijom glutaminske kiseline. Na Poljoprivrednoj eksperimentalnoj stanici u Conekticutu detaljan pregled biljnih proteina sastavio je Thomas Burr Osborne. Radeći sa Lafayette Mendel i primjenom Liebigovog zakona minimuma u hranjenju laboratorijskih miševa, ustanovljene su nutritivno esencijalne aminokiseline. Rad je nastavio i komunicirao William Cumming Rose. Razumijevanje proteina kao polipeptida došli su Franz Hofmeister i Hermann Emil Fischer u 1902.[12][13] Centralna uloga proteina kao enzima u živim organizmima nije bila u potpunosti cijenjena sve do 1926. godine, kada je James B. Sumner pokazao da je enzim ureaza u stvari protein.[14]
Poteškoće u pročišćavanju proteina u velikim količinama učinile su ih veoma teškim za proučavanje ranim proteinskim biohemičarima. Stoga su se rane studije fokusirale na proteine koji se mogu pročistiti u velikim količinama, naprimjer, krv, bjelance, razni toksini i probavne/metabolički enzimi dobiveni iz klaonica. Tokom 1950-ih, Armour Hot Dog Co. je pročistio 1 kg čiste goveđe pankreasne ribonukleaze A i učinio je slobodno dostupnom naučnicima; ovaj gest pomogao je da ribonukleaza A postane glavna meta biohemijskih studija u narednim decenijama.[7]
Linus Pauling je zaslužan za uspješno predviđanje sekundarne strukture regularnih proteina, zasnovane na vodikovim vezama, ideji koju je prvi iznio William Astbury 1933.[15] Kasniji rad Waltera Kauzmanna o denaturaciji,[16][17][18] zasnovano dijelom na prethodnim studijama Kaja Linderstrøm-Langa,[18] doprinijelo je razumijevanju svijanja proteina i strukture posredovane hidrofobnim interakcijama.
Prvi protein koji je sekvenciran bio je insulin, što je uradio Frederick Sanger, 1949. Sanger je ispravno odredio aminokiselinsku sekvencu insulina, čime je konačno pokazao da se proteini sastoje od linearnih polimera aminokiselina, a ne razgranatih lanaca, koloidaili citosola.[19] Za ovo dostignuće Dobio je Nobelovu nagradu, 1958.[20]

Sa razvojem rendgenske kristalografije, postalo je moguće sekvencirati proteinske strukture.[21] Prve strukture proteina koje je trebalo riješiti su hemoglobin, što je uradio Max Perutz, a Sir John Cowdery Kendrew, mioglobina, 1958.[22][23] Upotreba računara i povećanje računarske snage također su podržali sekvenciranje složenih proteina. Godine 1999. Roger Kornberg je uspio da sekvencira veoma složenu strukturu RNK-polimeraze, koristeći rendgenske zrake visokog intenziteta iz sinhrotrona.[21]
Od tada je razvijena krio-elektronska mikroskopija velikih makromolekulskih sklopova.[18]. Cryo-EM koristi uzorke proteina koji su zamrznuti, a ne kristale, i snopove elektrona, umjesto rendgenskih zraka. Prouzrokuje manje štete na uzorku, omogućavajući da se dobije više informacija i analiziraju veće strukture. Računarsko predviđanje strukture proteina malih proteina domena[18] je također pomogao istraživačima da pristupe rezoluciji proteinskih struktura na atomskom nivou. Od 2017., proteinska banka podataka ima preko 126.060 struktura proteina atomske rezolucije.[24]
Broj proteina kodiranih u genomima
[uredi | uredi izvor]Broj proteina kodiranih u genomu otprilike odgovara broju gena (iako može postojati značajan broj gena koji kodiraju RNK proteina, npr. ribosomske RNK). Virusi tipski kodiraju nekoliko do nekoliko stotina proteina, Archaea i bakterije nekoliko stotina do nekoliko hiljada, dok eukarioti tipski kodiraju nekoliko hiljada do desetina hiljada proteina (pogledajte veličina genoma za listu primjera).
Biohemija
[uredi | uredi izvor]

Većina proteina sastoji se od linearnih polimera, izgrađenih od niza do 20 različitih L-α- aminokiselina. Sve proteinogene aminokiseline imaju zajedničke strukturne karakteristike, uključujući α-ugljik na koji su vezane amino grupa, karboksil grupa i varijabilni bočni lanac. Samo prolin se razlikuje od ove osnovne strukture jer sadrži neobičan prsten za aminsku grupu N-kraja, koji tjera CO–NH amidni dio u fiksnu konformaciju.[25] Bočni lanci standardne aminokiseline, detaljno navedene u listi standardnih aminokiselina, imaju veliki izbor hemijskih struktura i svojstava; to je kombinovani efekat svih bočnih lanaca aminokiselina u proteinu koji na kraju određuje njegovu trodimenzijsku strukturu i njegovu hemijsku reaktivnost.[26]
Aminokiseline u polipeptidnom lancu povezane su peptidnom vezom. Kada se jednom poveže u proteinski lanac, pojedinačna aminokiselina naziva se „ostatak“, a povezani niz atoma ugljika, dušika i kisika poznati su kao „glavni lanac“ ili „proteinska kičma“.[27]:19
Peptidna veza ima dva rezonantna oblika koji doprinose nekom karakteru dvostruke veze i inhibiraju rotaciju oko svoje ose, tako da su alfa-ugljici otprilike komplanarni. Druga dva diedarna ugla u peptidnoj vezi određuju lokalni oblik koji preuzima proteinska kičma.[27]:31 Kraj sa slobodnom amino grupom poznat je kao N-kraj ili amino terminus, dok je kraj proteina sa slobodnom karboksilnom grupom poznat kao C-kraj ili karboksi kraj (sekvenca proteina je napisana od N-terminusa do C-terminus, s lijeva na desno).
Riječi protein, polipeptid i peptid su malo dvosmislene i mogu se preklapati u značenju. "Protein" se općenito koristi za označavanje kompletne biološke molekule u stabilnoj konformaciji, dok je "peptid" općenito rezerviran za kratke oligomere aminokiselina kojima često nedostaje stabilna 3D struktura. Ali granica između njih dvoje nije dobro definirana i obično je blizu 20-30 ostataka.[28] Polipeptid se može odnositi na bilo koji pojedinačni linearni lanac aminokiselina, obično bez obzira na dužinu, ali često implicira odsustvo definirane konformacije.
Interakcije
[uredi | uredi izvor]Proteini mogu komunicirati s mnogim tipovima molekula, uključujući sa drugim proteinima, lipidima, ugljikohidratima i sa DNK.[27][29][30][31]
Zastupljenost u ćelijama
[uredi | uredi izvor]Procijenjeno je da prosječne bakterije sadrže oko dva miliona proteina po ćeliji (npr. Escherichia coli i Staphylococcus aureus). Manje bakterije, kao što su Mycoplasma ili Spirochaeta sadrže manje molekula, reda veličine od 50.000 do jedan milion. Nasuprot tome, eukariotske ćelije su veće i stoga sadrže mnogo više proteina. Naprimjer, procjenjuje se da ćelije kvasca Saccharomyces cerevisiae sadrže oko 50 miliona proteina i ljudske ćelije reda veličine 1–3 milijarde.[32] Koncentracija pojedinačnih kopija proteina kreće se od nekoliko molekula do 20 miliona po ćeliji.[33] Nisu svi geni koji kodiraju proteine izraženi u većini ćelija i njihov broj zavisi od, naprimer, tipa ćelije i spoljašnjih podražaja. Naprimjer, od oko 20.000 proteina koje kodira ljudski genom, samo 6.000 je otkriveno u limfoblastoidnim ćelijama.[34]
Sinteza
[uredi | uredi izvor]Biosinteza
[uredi | uredi izvor]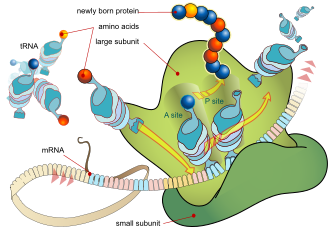

Proteini se sastavljaju od aminokiselina, koristeći informacije kodirane u genima. Svaki protein ima svoju jedinstvenu sekvencu aminokiselina koja je specificirana nukleotidnom sekvencom gena koji kodira ovaj protein. Genetički kod je skup skupova od tri nukleotida koji se nazivaju kodoni i svaka kombinacija od tri nukleotida označava aminokiselinu, na primjer AUG (adenin–uracil–guanin) je šifra za metionin. Pošto DNK sadrži četiri nukleotida, ukupan broj mogućih kodona je 64; dakle, postoji određena redundantnost u genetičkom kodu, sa nekim aminokiselinama specificiranim sa više od jednog kodona.[31]:1002–42 Geni kodirani u DNK su prvi transkribovani u preiRNK (iRNK) proteinima kao što je RNK polimeraza. Većina organizama zatim obrađuje pre-iRNK (također poznatu kao primarni transkript) koristeći različite oblike posttranslacijskih modifikacija kako bi formirali zrelu iRNK, koja se zatim koristi kao šablon za sintezu proteina u ribosomima. Kod prokariota, iRNK može se koristiti ili čim se proizvede, ili može biti vezana ribosomom nakon što se udalji od nukleoida. Nasuprot tome, eukarioti stvaraju iRNK u ćelijskom jedru, a zatim translociraju je preko jedarne membrane u citoplazmu, gdje se tada odvija sinteza proteina. Brzina sinteze proteina je veća kod prokariota nego kod eukariota i može doseći do 20 aminokiselina u sekundi.[35] Proces sintetizacije proteina iz iRNK šablona poznat je kao translacija. iRNK se učitava na ribosom i čita tri nukleotida u isto vrijeme, tako što se svaki kodon uparuje s njegovim baznim parom antikodona koji se nalazi na molekuli tRNK, koja nosi odgovarajuću aminokiselinu na kodon koji prepoznaje. Enzim aminoacil tRNK sintetaza "puni" molekule tRNK ispravnim aminokiselinama. Polipeptid koji raste često se naziva "nastajući lanac". Proteini se uvijek biosintetiziraju od N-kraja do C-kraja.[31]:1002–42
Veličina sintetiziranog proteina može se mjeriti brojem aminokiselina koje sadrži i njegovom ukupnom molekulskom masom, koja se obično iskazuje u jedinicama daltona (sinonim za jedinica atomske mase), ili jedinica derivata kilodalton (kDa). Prosječna veličina proteina raste od arheja preko bakterija do eukariota (283, 311, 438, odnosno 31, 34, 49 kDa ostataka) zbog većeg broja proteinskih domena koji čine proteine u višim organizmima.[36] Naprimjer, kvaščevi proteini su u prosjeku dugi 466 aminokiselina i 53 kDa u masi.[28] Najveći poznati proteini su titini, komponenta mišićne sarkomere, sa molekulskom masom od skoro 3.000 kDa i ukupnom dužinom od skoro 27.000 aminokiselina.[37]
Hemijska sinteza
[uredi | uredi izvor]Kratki proteini se takođe mogu sintetizirati hemijski pomoću porodice metoda poznatih kao sinteza peptida, koje se oslanjaju na organsku sintezu, tehnike kao što je hemijska ligacija za proizvodnju peptida u velikom prinosu.[38] Hemijska sinteza omogućava uvođenje neprirodnih aminokiselina u polipeptidne lance, kao što je vezivanje fluorescentnih sondi na bočne lance aminokiselina.[39] Ovi metodi su korisni u biohemijskim i citološkoj aboratoriji, iako općenito nisu za komercijalne primjene. Hemijska sinteza je neefikasna za polipeptide duže od oko 300 aminokiselina, a sintetirani proteini možda neće lakko preuzeti svoju nativnu tercijarnu strukturu. Većina metoda hemijske sinteze ide od C– do N-kraja, suprotno biološkoj reakciji.[40]
Struktura
[uredi | uredi izvor]

Lijevo: Reprezentacija svih atoma obojena tipom atoma.
Sredina: Pojednostavljeni prikaz koji ilustruje konformaciju okosnice, obojen sekundarnom strukturom.
Desno: Prikaz površine pristupačne rastvaraču obojen tipom ostatka (kiseli ostaci crveni, osnovni ostaci plavi, polarni ostaci zeleni, nepolarni ostaci bijeli).
Većina proteina savija se u jedinstvene 3D strukture. Oblik u koji se protein prirodno savija poznat je kao njegova nativna konformacija.[27]:36 Iako se mnogi proteini mogu savijati bez pomoći, jednostavno po hemijskim svojstvima njihovih aminokiselina, druge zahtijevaju pomoć molekulskih šaperona da se preklope u svoja nativna stanja.[27]:37 Biohemičari često govore o četiri različita aspekta struktura proteina:[27]:30–34
- Primarna struktura: aminokiselinska sekvenca. Protein je poliamid.
- Sekundarna struktura: lokalne strukture koje se redovno ponavljaju stabilizovane vodikovim vezama. Najčešći primjeri su α-heliks, β-list i okretanje. Budući da su sekundarne strukture lokalne, mnoge regije različite sekundarne strukture mogu biti prisutne u istoj proteinskoj molekuli.
- Tercijarna struktura: ukupni oblik jedne proteinske molekule; prostorni odnos sekundarnih struktura jedne prema drugoj. Tercijarna struktura se općenito stabilizuje nelokalnim interakcijama, najčešće formiranjem hidrofobnog jezgra, ali i preko slanih mostova, vodikove veze, disulfidne veze i čak i posttranslacijskih modifikacija. Termin "tercijarna struktura" koristi se često kao sinonim za termin "nabor". Tercijarna struktura je ono što kontrolira osnovnu funkciju proteina.
- Kvaternarna struktura: struktura formirana od nekoliko proteinskih molekula (polipeptidnih lanaca), obično nazvanih proteinske podjedinice u ovom kontekstu, koje funkcionišu kao jedan proteinski kompleks.
- Kvinarna struktura: potpisi površine proteina koji organizuju pretrpanu ćelijsku unutrašnjost. Kvinarna struktura ovisi o prolaznim, ali bitnim, makromolekulskim interakcijama koje se javljaju unutar živih ćelija.
Proteini nisu u potpunosti krute molekule. Pored ovih nivoa strukture, proteini se mogu prebacivati između nekoliko povezanih struktura dok obavljaju svoje funkcije. U kontekstu ovih funkcionalnih preuređivanja, ove tercijarne ili kvartarnarne strukture se obično nazivaju konformacije, a prijelazi između njih se nazivaju "konformacijske promjene". Takve promjene često su izazvane vezivanjem molekula supstrata do aktivnog mjesta enzima, ili fizičkog regiona proteina koji učestvuje u hemijskoj katalizi. U rastvoru, proteini takođe prolaze kroz varijacije u strukturi usljed termičkih vibracija i sudara sa drugim molekulama.[31]:368–75

Proteini se mogu neformalno podijeliti u tri glavne klase, koje su u korelaciji sa tipskim tercijarnim strukturama: globulasti proteini, vlaknasti proteini, i membranski proteini. Gotovo svi globulasti proteini su topivi i mnogi su enzimi. Vlaknasti proteini su često strukturni, kao što je kolagen, glavna komponenta vezivnog tkiva, ili keratin, proteinska komponenta dlaka i noktiju. Membranski proteini često služe kao receptori ili obezbjeđuju kanale za prolazak polarnih ili nabijenih molekula kroz ćelijsku membranu.[31]:165–85
Poseban slučaj intramolekulskih vodikovih veza unutar proteina, koji su slabo zaštićeni od napada vode i stoga promovišu vlastitu dehidraciju, nazivaju se dehidroni.[41]
Proteinski domeni
[uredi | uredi izvor]Mnogi proteini sastoje se od nekoliko proteinskih domena, tj. segmenata proteina koji se savijaju u različite strukturne jedinice. Domeni obično također imaju specifične funkcije, kao što su enzimske aktivnosti (npr. kinaza) ili služe kao moduli vezivanja (npr. SH3-domen vezuje se za sekvence bogate prolinom u drugim proteinima) .
Motiv sekvence
[uredi | uredi izvor]Kratke sekvence aminokiselina unutar proteina često djeluju kao mjesta prepoznavanja za druge proteine.[42] Naprimjer, SH3 domen se obično vezuje za kratke PxxP motive (tj. dva prolina [P], odvojena sa dvije nespecificirane aminokiseline [x], iako okolne aminokiseline mogu odrediti tačnu specifičnost vezivanja). Mnogi takvi motivi prikupljeni su u bazi podataka Eukaryotic Linear Motif (ELM).
Ćelijske funkcije
[uredi | uredi izvor]Proteini su glavni akteri unutar ćelije, za koje se kaže da izvršavaju funkcije određene informacijama kodiranim u genima.[28] Sa izuzetkom određenih tipova RNK, većina drugih bioloških molekula je relativno inertnih elemenata na koje djeluju proteini. Proteini čine polovinu suhe težine ćelije Escherichia coli, dok druge makromolekule kao što su DNK i RNK čine samo 3% odnosno 20%.[43] Skup proteina izraženih u određenoj ćeliji ili tipu ćelije poznat je kao njegov proteom.

Glavna karakteristika proteina koja također omogućava njihov raznolik skup funkcija je njihova sposobnost da specifično i čvrsto vežu druge molekule. Područje proteina odgovornog za vezivanje drugog molekula poznato je kao mjesto vezivanja i često je udubljenje ili "džep" na površini molekula. Ova sposobnost vezivanja je posredovana tercijarnom strukturom proteina, koja definira džep za mjesto vezivanja, i hemijskim svojstvima bočnih lanaca okolnih aminokiselina. Vezivanje za proteine može biti izuzetno čvrsto i specifično; naprimjer, protein inhibitor ribonukleaze veže se za ljudski angiogenin sa subfemtomolarnom konstantom disocijacije (<10−15 M) ali uopće ne veže na njegov homolog onkonaza vodozemaca (>1 M). Ekstremno male hemijske promene, kao što je dodavanje jedne metil grupe vezujućem partneru ponekad mogu biti dovoljne da se skoro eliminiše vezivanje; naprimjer, aminoacil tRNK sintetaza specifična za aminokiselinu valin diskriminira vrlo sličan bočni lanac aminokiseline izoleucin.[44]
Proteini se mogu vezati za druge proteine kao i za malomolekulske supstrate. Kada se proteini specifično vežu za druge kopije iste molekule, mogu se oligomerizirati i formirati u fibrile; ovaj proces se često dešava u strukturnim proteinima koji se sastoje od globulastih monomera koji se samopovezuju i formiraju kruta vlakna. Interakcije protein-protein također reguliraju enzimsku aktivnost, kontroliraju napredovanje kroz ćelijski ciklus i omogućavaju sklapanje velikih proteinskih kompleksa koji izvode mnoge blisko povezane reakcije sa zajedničkom biološkom funkcijom . Proteini se također mogu vezati ili čak integrirati u ćelijske membrane. Sposobnost partnera u vezivanju da izazovu konformacijske promjene u proteinima omogućava izgradnju enormno složenih mreža signalizacija.[31]:830–49
Kako su interakcije između proteina reverzibilne i u velikoj mjeri zavise od dostupnosti različitih grupa proteina partnera za formiranje agregata koji su sposobni da obavljaju diskretne skupove funkcija, proučavanje interakcija između specifičnih proteina je ključ za razumijevanje važnih aspekata ćelijske funkcije, i konačno svojstava koja razlikuju određene tipove ćelija.
Enzimi
[uredi | uredi izvor]Najpoznatija uloga proteina u ćeliji je kao enzima, koji katalizuju hemijske reakcije. Enzimi su obično vrlo specifični i ubrzavaju samo jednu ili nekoliko hemijskih reakcija. Enzimi izvode većinu reakcija uključenih u metabolizam, kao i manipulišu DNK u procesima kao što su DNK replikacija, popravka DNK i transkripcija. Neki enzimi djeluju na druge proteine kako bi dodali ili uklonili hemijske grupe u procesu poznatom kao posttranslacijska modifikacija. Poznato je da oko 4.000 reakcija kataliziraju enzimi.[45] Ubrzanje brzine enzimske katalize je često enormno – čak 1017-puta povećanje brzine u odnosu na nekataliziranu reakciju u slučaju orotat-dekarboksilaza (78 miliona godina bez enzima, 18 milisekundi sa enzimom).[46]
Vezane molekule i na koje djeluju enzimi nazivaju se supstrati. Iako se enzimi mogu sastojati od stotina aminokiselina, obično je samo mali dio ostataka koji dolazi u kontakt sa supstratom, a još manji dio – u prosjeku tri do četiri ostatka – koji su direktno uključeni u katalizu.[47] Područje enzima koji veže supstrat i sadrži katalitske ostatke poznato je kao aktivno mjesto. Dirigentni proteini su članovi klase proteina koji diktiraju stereohemijska svojstva spojeva sintetiziranih drugim enzimima.[48]
Ćelijska signalizacija i vezivanje liganda
[uredi | uredi izvor]
Mnogi proteini uključeni su u proces ćelijske signalizacije i transdukcije signala. Neki proteini, kao što je insulin, su vanćelijskii proteini koji prenose signal iz ćelije u kojoj su sintetizovani do drugih ćelija u udaljenim biološkim tkivima. Drugi su membranski proteini koji djeluju kao receptori. čija je glavna funkcija vezati signalnu molekulu i izazvati biohemijski odgovor u ćeliji. Mnogi receptori imaju mjesto vezivanja izloženo na površini ćelije i efektorski domen unutar ćelije, koji može imati enzimsku aktivnost ili može biti podvrgnut konformacijskoj promjeni koju detektuju drugi proteini unutar ćelije.[30]:251–81
Antitijela su proteinske komponente prilagodljivog imunskog sistema čija je glavna funkcija da vežu antigene ili strane supstance u tijelu, i ciljaju ih za uništenje. Antitijela se mogu izlučiti u vanćelijsko okruženje ili usidriti u membranama specijalizovanih B-ćelija poznatih kao plazmaćelije. Dok su enzimi ograničeni u svom afinitetu vezivanja za svoje supstrate neophodnošću sprovođenja njihove reakcije, antitijela nemaju takva ograničenja. Afinitet vezivanja antitijela za njegovu metu je izuzetno visok.[31]:275–50
Mnogi transportni proteini liganda vezuju određene male biomolekule i transportuju ih na druge lokacije u tijelu višećelijskog organizma. Ovi proteini moraju imati visok afinitet vezivanja kada je njihov ligand prisutan u visokim koncentracijama, ali također moraju oslobađati ligand kada je prisutan u niskim koncentracijama u ciljnom tkivu. Kanonski primjer proteina koji veže ligand je hemoglobin, koji prenosi kisik iz pluća u druge organe i tkiva u svim kičmenjacima i ima bliske homologe u svakom biološkom carstvu.[31]:222–29 Lektini su proteini koji vezuju šećer, vrlo specifični po svojim šećernim dijelovima. Lektini obično imaju ulogu u biološkim fenomenima prepoznavanja koji uključuju ćelije i proteine.[49] Receptori i hormoni su visoko specifičnih vezujući proteini.
Transmembranski proteini također mogu poslužiti kao transportni proteini liganda koji mijenjaju propusnost ćelijske membrane za male molekule i ione. Sama membrana ima hidrofob nojezgro kroz koje polarne ili nabijene molekule ne mogu difundirati. Membranski proteini sadrže unutrašnje kanale koji omogućavaju takvim molekulima da uđu i izađu iz ćelije. Mnogi ionski kanalni proteini su specijalizovani za odabir samo za određeni ion; naprimjer, kalijski i natrijski kanali često diskriminiraju samo jedan od dva jona.[30]:232–34
Strukturni proteini
[uredi | uredi izvor]Strukturni proteini daju krutost i čvrstoću inače tekućim biološkim komponentama. Većina strukturnih proteina su vlaknasti proteini; naprimjer, kolagen i elastin su kritične komponente vezivnog tkiva kao što je hrskavica, a keratin se nalazi u tvrdim ili filamentnim strukturama kao što su dlaka, nokti, perje, papci i neke životinjske školjke.[31]:178–81 Neki globulasti proteini također mogu imati strukturne funkcije, naprimjer, aktin i tubulin su globulasti i rastvorljivi kao monomeri, ali polimeri se formiraju u duga, čvrsta vlakna koja čine citoskelet, što omogućava ćeliji da zadrži svoj oblik i veličinu.
Ostali proteini koji služe strukturnim funkcijama su motorni protein kao što su miozin, kinezin i dinein, koji su sposobni da generišu mehaničke sile. Ovi proteini su ključni za ćelijski motilitet jednoćelijskih organizama i spermatozoida mnogih višećelijskih organizama koji se razmnožavaju seksualno. Oni također stvaraju sile koje se stvaraju kontrakcijom mišića[31]:258–64, 272 i imaju bitnu ulogu u unutarćelijskom transportu.
Evolucija proteina
[uredi | uredi izvor]Ključno pitanje u molekulskoj biologiji je kako proteini evoluiraju, tj. kako mutacije (ili bolje rečeno promjene u aminokiselinskoj sekvenci) mogu dovesti do novih struktura i funkcija? Većina aminokiselina u proteinu može se promijeniti bez ometanja aktivnosti ili funkcije, kao što se može vidjeti iz brojnih homolognih proteina u različitim vrstama (sakupljenih u specijalizovanim bazama podataka za porodice proteina, npr. PFAM).[50] Kako bi se spriječile dramatične posljedice mutacija, gen može biti dupliciran prije nego što može slobodno mutirati. Međutim, ovo također može dovesti do potpunog gubitka funkcije gena, a time i pseudogena.[51] Češće, promjene jedne aminokiseline (tačkasta mutacija) imaju ograničene posljedice, iako neke mogu značajno promijeniti funkciju proteina, posebno u enzimima. Naprimjer, mnogi enzimi mogu promijeniti svoju specifičnost supstrata jednom ili nekoliko mutacija.[52] Promjene u specifičnosti supstrata su olakšane promiskuitetom supstrata, tj. sposobnošću mnogih enzima da vežu i obrađuju više supstrata. Kada se pojave mutacije, specifičnost enzima može se povećati (ili smanjiti), a time i njegova enzimska aktivnost.[52] Tako se bakterije (ili drugi organizmi) mogu prilagoditi različitim izvorima hrane, uključujući neprirodne supstrate kao što su plastični.[53]
Metodi proučavanja
[uredi | uredi izvor]Aktivnosti i strukture proteina mogu se ispitati in vitro, in vivo, i in silico. In vitro studije pročišćenih proteina u kontroliranim sredinama korisne su za spoznaju kako protein obavlja svoju funkciju: naprimjer, studije kinetike enzima istražuju hemijski mehanizam katalitske aktivnosti enzima i njegovog relativnog afiniteta za različite moguće molekule supstrata. Nasuprot tome, 'in vivo' eksperimenti mogu pružiti informacije o fiziološkoj ulozi proteina u kontekstu ćelije ili čak cijelog organizma. 'In silico' studije koriste računarske metode za proučavanje proteina.
Prečišćavanje proteina
[uredi | uredi izvor]Da bi se izvršila in vitro analiza, protein mora biti prečišćen od drugih ćelijskih komponenti. Ovaj proces obično počinje lizom ćelija, pri čemu se ćelijska membrana poremeti i njen unutrašnji sadržaj se oslobađa u rastvor poznat kao sirovi lizat. Rezultirajuća smjesa se može pročistiti pomoću ultracentrifugacije, koja frakcioniše različite ćelijske komponente u frakcije koje sadrže rastvorljive proteine; membranski lipidi i proteini, ćelijske organele i nukleinske kiseline. Precipitacija metodom poznatim kao soljenje može koncentrirati proteine iz ovog lizata. Različiti tipovi hromatografija se zatim koriste za izolaciju proteina ili proteina od interesa, na osnovu svojstava kao što su molekulskaa težina, neto naboj i afinitet vezivanja.[27]:21–24 Nivo pročišćavanja može se pratiti korištenjem različitih tipova gel elektroforeza, ako su poznati molekulska težina željenog proteina i izoelektrična tačka, spektroskopijom ako protein ima prepoznatljive spektroskopske karakteristike, ili pomoću enzimskih testova ako protein ima enzimsku aktivnost. Dodatno, proteini se mogu izolovati prema njihovom naboju pomoću elektrofokusiranje.[54] Za prirodne proteine može biti potreban niz koraka prečišćavanja kako bi se dobio protein dovoljno čist za laboratorijske primjene. Da bi se pojednostavio ovaj proces, se često koristi genetičko inženjerstvo, za dodavanje hemijskih karakteristika proteinima koje ih čine lakšim za pročišćavanje bez uticaja na njihovu strukturu ili aktivnost. Ovdje je "oznaka" koja se sastoji od specifične aminokiselinske sekvence, često niza histidinskih ostataka ("His-tag"), vezana za jedan kraj proteina. Kao rezultat toga, kada se lizat prođe preko hromatografske kolone koja sadrži nikl, ostaci histidina povezuju nikl i vezuju se za kolonu, dok neoznačene komponente lizata prolaze nesmetano. Razvijeno je više različitih oznaka koje pomažu istraživačima da pročiste specifične proteine iz složenih mješavina.[55]
Ćelijska lokalizacija
[uredi | uredi izvor]
Proučavanje proteina "in vivo" često se bavi sintezom i lokalizacijom proteina unutar ćelije. Iako se mnogi unutarćelijski proteini sintetiziraju u citoplazmi postoje i proteini vezani za membranu ili se izlučuju u endoplazmatskom retikulumu, specifičnosti načina na koji su proteini ciljani na određene organele ili ćelijske strukture su često nejasne. Korisna tehnika za procjenu ćelijske lokalizacije koristi genetičko enjerstvo za ekspresiju u ćeliji fuzijskih proteina ili himera, koja se sastoji od prirodnog proteina od interesa povezanog sa "reporterrom" kao što je zeleni fluorescentni protein (GFP).[56] Položaj spojenog proteina unutar ćelije može se jasno i efikasno vizualizirati pomoću mikroskopije,[57] kao što je prikazano na suprotnoj slici.
Ostali metodi za razjašnjavanje ćelijske lokacije proteina zahtijevaju korištenje poznatih kompartmentnih markera za regije kao što su ER, Golgijev aparat, lizosomi ili vakuole, mitohondrije, hloroplasti, plazmamembrana, itd. Uz upotrebu fluorescentno označenih verzija ovih markera ili antitijela na poznate markere, postaje mnogo jednostavnije identificirati lokalizaciju proteina od interesa. Naprimjer, indirektna imunofluorescencija će omogućiti kolokalizaciju fluorescencije i demonstraciju lokacije. Za označavanje ćelijskih odjeljaka za sličnu svrhu koriste se fluorescentne boje.[58]
Postoje i druge mogućnosti. Naprimjer, imunohistohemija obično koristi antitijelo na jedan ili više proteina od interesa koji su konjugirani sa enzimima dajući ili luminiscentne ili hromogene signale koji se mogu porediti između uzoraka, omogućavajući informacije o lokalizaciji. Druga primjenjiva tehnika je kofrakcioniranje u gradijentima saharoze (ili drugog materijala), korištenjem izopikničkog centrifugiranja.[59] Iako ova tehnika ne dokazuje kolokalizaciju odjeljka poznate gustine i proteina od interesa, povećava se vjerovatnoća, i podložniji je studijama velikih razmjera.
Konačno, zlatni standard metoda ćelijske lokalizacije je imunoelektronska mikroskopija. Ova tehnika također koristi antitijelo na protein od interesa, zajedno sa klasičnim tehnikama elektronske mikroskopije. Uzorak se priprema za uobičajeno elektronsko mikroskopsko ispitivanje, a zatim se tretira antitijelom na protein od interesa, koje je konjugirano s izuzetno elektro-gustim materijalom, obično zlatom. Ovo omogućava lokalizaciju i ultrastrukturnih detalja kao i proteina od interesa.[60]
Kroz drugu primjenu genetičkog inženjerstva poznatu kao mjesno usmjerena mutageneza, istraživači mogu promijeniti sekvencu proteina, a time i njegovu strukturu, ćelijsku lokalizaciju i podložnost regulaciji. Ova tehnika čak dozvoljava inkorporaciju neprirodnih aminokiselina u proteine, koristeći modifikovane tRNK,[61] i može omogućiti racionalan dizajn novih proteina sa novim svojstvima.[62]
Proteomika
[uredi | uredi izvor]Ukupni komplement proteina prisutnih u jednom trenutku u ćeliji ili tipu ćelije poznat je kao njen proteom, a proučavanje takvih velikih skupova podataka definiše polje proteomika, nazvano po analogiji sa srodnaom oblasti genomika. Ključne eksperimentalne tehnike u proteomici uključuju 2D elektroforezu,[63] koja omogućava odvajanje mnogih proteina, masenu spektometriju,[64] koja omogućava brzu identifikaciju proteina visoke propusnosti i sekvenciranje peptida (najčešće nakon digestija u gelu), proteinske mikromreže, koji omogućavaju detekciju relativnih nivoa različitih proteina prisutnih u ćeliji, i dvo-hibridni skrining, koji omogućava sistematsko istraživanje interakcija protein-protein.[65] Ukupni komplement biološki mogućih takvih interakcija poznat je kao interaktom.[66] Sistematski pokušaj određivanja struktura proteina koji predstavljaju svaki mogući nabor poznat je kao strukturna genomika.[67]
Određivanje strukture
[uredi | uredi izvor]Otkrivanje tercijarne strukture proteina, ili kvaternarne strukture njegovih kompleksa, može pružiti važne naznake o tome kako protein obavlja svoju funkciju i kako na njega može uticati, naprimjer u dizajn lijekova. Kako su proteini premali da bi se vidjeli pod svjetlosnim mikroskopom, moraju se koristiti drugi metodi da bi se odredila njihova struktura. Uobičajene eksperimentalne metode uključuju rendgensku kristalografiju i NMR spektroskopiju, od kojih obje mogu proizvesti strukturne informacije pri atomskoj rezoluciji. Međutim, NMR eksperimenti mogu pružiti informacije iz kojih se može procijeniti podskup udaljenosti između parova atoma, a konačne moguće konformacije za protein se određuju rješavanjem problema geometrija udaljenosti. Interferometrija dvostruke polarizacije je kvantitativni analitički metod za mjerenje ukupne konformacije proteina i konformacijske promjene uzrokovane interakcijama ili drugim stimulusom. Kružni dihroizam je još jedna laboratorijska tehnika za određivanje unutrašnjeg β-list /α-helikalnog sastava proteina. Krioelektronska mikroskopija koristi se za proizvodnju strukturnih informacija niže rezolucije o veoma velikim proteinskim kompleksima, uključujući sastavljene viruse;[30]:340–41 varijanta poznata kao elektronska kristalografija takođe može proizvesti informacije visoke rezolucije u nekim slučajevima, posebno za dvodimenzijske kristale membranskih proteina.[68] Riješene strukture se obično deponuju u Proteinskoj banci podataka (PDB), besplatno dostupnom resursu iz kojeg se mogu dobiti strukturni podaci o hiljadama proteina u obliku Kartezijanskih koordinata za svaki atom u proteinu.[69]
Poznato je mnogo više genskih sekvenci od proteinskih struktura. Nadalje, skup riješenih struktura je pristrasan prema proteinima koji se lahko mogu podvrgnuti uvjetima potrebnim u X-zračnoj kristalografiji, jednom od glavnih metoda određivanja strukture. Konkretno, globulaste proteine je relativno lahko kristalizirati u pripremi za rendgensku kristalografiju. Membranske proteine i velike proteinske komplekse je, nasuprot tome, teško kristalizirati i nedovoljno su zastupljeni u PDB-u.[70] Strukturnogenomičke inicijative pokušale su ispraviti ove nedostatke sistematskim rješavanjem reprezentativnih struktura glavnih klasa nabora. Metodi predviđanja strukture proteina pokušavaju da obezbede sredstvo za generiranje verodostojne strukture za proteine čije strukture nisu eksperimentalno određene.[71]
Predviđanje strukture
[uredi | uredi izvor]
Komplementarno polju strukturne genomike, predikcija strukture proteina razvija efikasne matematičke modele proteina za kompjutersko predviđanje molekulskih formacija u teoriji, umjesto otkrivanja struktura uz laboratorijsko posmatranje.[72] Najuspješniji tip predviđanja strukture, poznat kao homološko modeliranje, oslanja se na postojanje strukture "šablona" sa sličnošću sekvence sa proteinom koji se modelira. Cilj strukturne genomike je osigurati dovoljnu zastupljenost u riješenim strukturama za modeliranje većine preostalih.[73] Iako proizvodnja tačnih modela ostaje izazov kada su dostupne samo udaljene strukture šablona, sugerirano je da je poravnavanje sekvence usko grlo u ovom procesu, jer se mogu proizvesti prilično precizni modeli ako je poznato "savršeno" poravnanje sekvence.[74] Mnogi metodi predviđanja strukture poslužili su da informišu novonastajuće polje proteinsko inženjerstvo, u kojem su već dizajnirani novi proteinski nabori.[75] Također proteini (kod eukariota ~33%) sadrže velike nestrukturirane, ali biološki funkcionalne segmente i mogu se klasifikovati kao intrinzično poremećeni proteini.[76] Predviđanje i analiza poremećaja proteina je, stoga, važan dio karakterizacije strukture proteina.[77]
Bioinformatika
[uredi | uredi izvor]Razvijen je širok spektar računarskih metoda za analizu strukture, funkcije i evolucije proteina. Razvoj takvih alata podstaknut je velikom količinom genomskih i proteomskih podataka dostupnih za razne organizme, uključujući ljudski genom. Prosto je nemoguće eksperimentalno proučiti sve proteine, pa se samo nekoliko podvrgava laboratorijskim eksperimentima dok se računarski alati koriste za ekstrapolaciju na slične proteine. Takvi homologni proteini mogu se efikasno identifikovati u udaljenim organizmima pomoću usklađivanja sekvenci. Genom i sekvence gena mogu se pretraživati raznim alatima za određena svojstva. Alat za profilisanje sekvenci mogu pronaći restrikcijski enzimi mesta, otvoreni okvir čitanja u nukleotidnim sekvencama i predvidjeti sekundarne strukture. Mogu se konstruirati filogenetska stabla i razvijati evolucijske hipoteze pomoću specijalnog softvera kao što je ClustalW u vezi sa porijeklom modernih organizama i genima koje oni izražavaju. Za analizu gena i proteina, sada je neophodna oblast bioinformatike.
In silico simulacija dinamičkih procesa
[uredi | uredi izvor]Složeniji računarski problem je predviđanje međumolekulskih interakcija, kao što su molekulsko spajanje,[78] preklapanje proteina, interakcija protein-protein i hemijska reaktivnost. Matematički modeli za simulaciju ovih dinamičkih procesa uključuju molekulsku mehaniku, posebno molekulsku dinamiku. S tim u vezi, in silico simulacije su otkrile savijanje malih α-heliksnih proteinskih domena kao što je villinsko pokrivalo,[79] HIV pomoćni protein[80] i hibridne metode koje kombinuju standardnu molekulsku dinamiku sa kvantnomehaničkom matematikom istražili su elektronska stanja rodopsina.[81]
Osim klasične molekulske dinamike, metodi kvantne dinamike omogućavaju simulaciju proteina u atomskim detaljima sa preciznim opisom kvantnomehaničkih efekata. Primjeri uključuju višeslojni višekonfiguracijski vremenski ovisni Hartree (MCTDH) metod i ) pristup hijerarhijskih jednačina kretanja (HEOM, koji su primijenjeni na kriptohrome biljaka[82] i kompleksi za prikupljanje svjetlosti bakterija.[83]. I kvantne i klasične mehaničke simulacije bioloških sistema su izuzetno računarski zahtjevne, tako da inicijative distribuiranog računarstva (naprimjer, Folding@home projekt[84]) olakšavaju molekulsko modeliranje, iskorištavanjem napretka u GPU paralelnom procesuiranju i Monte Carlo tehnikama.
Hemijska analiza
[uredi | uredi izvor]Ukupan sadržaj dušika u organskoj materiji uglavnom formiraju amino grupe u proteinima. Ukupni Kjeldahlov azot (TKN) je mjera dušika koja se široko koristi u analizi (otpadne) vode, tla, hrane, hrane za životinje i organske tvari općenito. Kao što naziv govori, primjenjuje se Kjeldahlova metoda. Dostupne su osjetljivije metode.[85][86]
Prehrana
[uredi | uredi izvor]Većina mikroorganizama i biljaka može biosintetizirati svih 20 standardnih aminokiselina, dok životinje (uključujući i ljude) moraju dobiti neke od aminokiselina iz ishrane.[43] Aminokiseline koje organizam ne može sam sintetizirati nazivaju se esencijalne aminokiseline. Ključni enzimi koji sintetiziraju određene aminokiseline nisu prisutni kod životinja – kao što je aspartokinaza, koja katalizira prvi korak u sintezi lizina, metionina i treonina iz aspartata. Ako su aminokiseline prisutne u okolini, mikroorganizmi mogu sačuvati energiju tako što preuzimaju aminokiseline iz svog okruženja i podreguliraju svoje biosintetske puteve.
Kod životinja se aminokiseline dobijaju konzumiranjem hrane koja sadrži proteine. Progutani proteini se zatim razlažu na aminokiseline putem probave, što obično uključuje denaturaciju proteina izlaganjem kiselinama i hidrolizi enzimima zvanim proteaze. Neke unesene aminokiseline koriste se za biosintezu proteina, dok se druge pretvaraju u glukozu putem glukoneogeneze, ili se unose u ciklus limunske kiseline. Ova upotreba proteina kao goriva je posebno važna u uslovima gladovanja jer omogućava da se sopstveni tjelesni proteini koriste za održavanje života, posebno oni koji se nalaze u mišićima.[87]
Kod životinja kao što su psi i mačke, proteini održavaju zdravlje i kvalitet kože podstičući rast folikula dlake i keratinizaciju i na taj način smanjujući vjerojatnost problema s kožom koji izazivaju neugodne mirise.[88] Proteini lošeg kvaliteta također imaju ulogu u zdravlju gastrointestinalnog trakta, povećavajući potencijal za nadimanje i neugodne spojeve kod pasa, jer kada proteini stignu do debelog crijeva u nesvarenom stanju, fermentiraju se proizvodeći plin sumporovodik, indol i skatol.[89] Psi i mačke bolje probavljaju životinjske proteine nego one iz biljaka, ali proizvodi životinjskog porijekla lošeg kvaliteta probavljaju se loše, uključujući kožu, perje i vezivno tkivo.[89]
Bjelančevine u ishrani
[uredi | uredi izvor]Bjelančevine se nalaze u raznim vrstama prehrambenih namirnica. Može se gotovo reći da su u većim ili manjim količinama zastupljeni u svoj hrani osim u rafiniranim šećerima i mastima. Hrana životinjskog porijekla poput mesa, riba, jaja, mlijeka, jogurta i sira dobar su izvor proteina u kvalitativnom i kvantitativnom smislu. Osim što sadrže mnogo proteina te su namirnice izvor svih esencijalnih aminokiselina.
Također pogledajte
[uredi | uredi izvor]Reference
[uredi | uredi izvor]- ^ Hadžiselimović R., Pojskić N. (2005): Uvod u humanu imunogenetiku. Institut za genetičko inženjerstvo i biotehnologiju (INGEB), Sarajevo, ISBN 9958-9344-3-4.
- ^ Kapur Pojskić L., Ed. (2014): Uvod u genetičko inženjerstvo i biotehnologiju, 2. izdanje. Institut za genetičko inženjerstvo i biotehnologiju (INGEB), Sarajevo, ISBN 978-9958-9344-8-3.
- ^ Kornberg A. (1989): For the love of enzymes – The Odyssay of a biochemist. Harvard University Press, Cambridge (Mass.), London, ISBN 0-674-30775-5, ISBN 0-674-30776-3.
- ^ Thomas Burr Osborne (1909): The Vegetable Proteins Arhivirano 22. 3. 2016. na Wayback Machine, History pp 1 to 6, from archive.org
- ^ Mulder GJ (1838). "Sur la composition de quelques substances animales". Bulletin des Sciences Physiques et Naturelles en Néerlande: 104.
- ^ Harold H (1951). "Origin of the Word 'Protein.'". Nature. 168 (4267): 244. Bibcode:1951Natur.168..244H. doi:10.1038/168244a0. PMID 14875059. S2CID 4271525.
- ^ a b c Perrett D (august 2007). "From 'protein' to the beginnings of clinical proteomics". Proteomics: Clinical Applications. 1 (8): 720–38. doi:10.1002/prca.200700525. PMID 21136729. S2CID 32843102.
- ^ New Oxford Dictionary of English
- ^ Reynolds JA, Tanford C (2003). Nature's Robots: A History of Proteins (Oxford Paperbacks). New York, New York: Oxford University Press. str. 15. ISBN 978-0-19-860694-9.
- ^ Reynolds and Tanford (2003).
- ^ Bischoff TL, Voit C (1860). Die Gesetze der Ernaehrung des Pflanzenfressers durch neue Untersuchungen festgestellt (jezik: njemački). Leipzig, Heidelberg.
- ^ "Hofmeister, Franz". encyclopedia.com. Arhivirano s originala, 5. 4. 2017. Pristupljeno 4. 4. 2017.
- ^ "Protein, section: Classification of protein". britannica.com. Arhivirano s originala, 4. 4. 2017. Pristupljeno 4. 4. 2017.
- ^ Sumner JB (1926). "The isolation and crystallization of the enzyme urease. Preliminary paper" (PDF). Journal of Biological Chemistry. 69 (2): 435–41. doi:10.1016/S0021-9258(18)84560-4. Arhivirano s originala, 25. 3. 2011. Pristupljeno 16. 1. 2011.
- ^ Pauling L, Corey RB (maj 1951). "Atomic coordinates and structure factors for two helical configurations of polypeptide chains" (PDF). Proceedings of the National Academy of Sciences of the United States of America. 37 (5): 235–40. Bibcode:1951PNAS...37..235P. doi:10.1073/pnas.37.5.235. PMC 1063348. PMID 14834145. Arhivirano (PDF) s originala, 28. 11. 2012. Pristupljeno 14. 4. 2009.
- ^ Kauzmann W (maj 1956). "Structural factors in protein denaturation". Journal of Cellular Physiology. 47 (Suppl 1): 113–31. doi:10.1002/jcp.1030470410. PMID 13332017.
- ^ Kauzmann W (1959). "Some factors in the interpretation of protein denaturation". Advances in Protein Chemistry Volume 14. Advances in Protein Chemistry. 14. str. 1–63. doi:10.1016/S0065-3233(08)60608-7. ISBN 978-0-12-034214-3. PMID 14404936.
- ^ a b c d Kalman SM, Linderstrøm-Lang K, Ottesen M, Richards FM (februar 1955). "Degradation of ribonuclease by subtilisin". Biochimica et Biophysica Acta. 16 (2): 297–99. doi:10.1016/0006-3002(55)90224-9. PMID 14363272.
- ^ Sanger F (1949). "The terminal peptides of insulin". The Biochemical Journal. 45 (5): 563–74. doi:10.1042/bj0450563. PMC 1275055. PMID 15396627.
- ^ Sanger F. (1958), Nobel lecture: The chemistry of insulin (PDF), Nobelprize.org, arhivirano (PDF) s originala, 19. 3. 2013, pristupljeno 9. 2. 2016
- ^ a b Stoddart, Charlotte (1. 3. 2022). "Structural biology: How proteins got their close-up". Knowable Magazine. doi:10.1146/knowable-022822-1. Pristupljeno 25. 3. 2022.
- ^ Muirhead H, Perutz MF (august 1963). "Structure of hemoglobin. A three-dimensional fourier synthesis of reduced human hemoglobin at 5.5 Å resolution". Nature. 199 (4894): 633–38. Bibcode:1963Natur.199..633M. doi:10.1038/199633a0. PMID 14074546. S2CID 4257461.
- ^ Kendrew JC, Bodo G, Dintzis HM, Parrish RG, Wyckoff H, Phillips DC (mart 1958). "A three-dimensional model of the myoglobin molecule obtained by x-ray analysis". Nature. 181 (4610): 662–66. Bibcode:1958Natur.181..662K. doi:10.1038/181662a0. PMID 13517261. S2CID 4162786.
- ^ "RCSB Protein Data Bank". Arhivirano s originala, 18. 4. 2015. Pristupljeno 19. 1. 2017.
- ^ Nelson DL, Cox MM (2005). Lehninger's Principles of Biochemistry (4th izd.). New York, New York: W. H. Freeman and Company.
- ^ Gutteridge A, Thornton JM (novembar 2005). "Understanding nature's catalytic toolkit". Trends in Biochemical Sciences. 30 (11): 622–29. doi:10.1016/j.tibs.2005.09.006. PMID 16214343.
- ^ a b c d e f g Murray RF, Harper HW, Granner DK, Mayes PA, Rodwell VW (2006). Harper's Illustrated Biochemistry. New York: Lange Medical Books/McGraw-Hill. ISBN 978-0-07-146197-9.
- ^ a b c Lodish H, Berk A, Matsudaira P, Kaiser CA, Krieger M, Scott MP, Zipurksy SL, Darnell J (2004). Molecular Cell Biology (5th izd.). New York, New York: WH Freeman and Company.
- ^ Ardejani MS, Powers ET, Kelly JW (august 2017). "Using Cooperatively Folded Peptides To Measure Interaction Energies and Conformational Propensities". Accounts of Chemical Research. 50 (8): 1875–1882. doi:10.1021/acs.accounts.7b00195. PMC 5584629. PMID 28723063.
- ^ a b c d Branden C, Tooze J (1999). Introduction to Protein Structure. New York: Garland Pub. ISBN 978-0-8153-2305-1.
- ^ a b c d e f g h i j Van Holde KE, Mathews CK (1996). Biochemistry. Menlo Park, California: Benjamin/Cummings Pub. Co., Inc. ISBN 978-0-8053-3931-4.
- ^ Milo R (decembar 2013). "What is the total number of protein molecules per cell volume? A call to rethink some published values". BioEssays. 35 (12): 1050–55. doi:10.1002/bies.201300066. PMC 3910158. PMID 24114984.
- ^ Beck M, Schmidt A, Malmstroem J, Claassen M, Ori A, Szymborska A, Herzog F, Rinner O, Ellenberg J, Aebersold R (novembar 2011). "The quantitative proteome of a human cell line". Molecular Systems Biology. 7: 549. doi:10.1038/msb.2011.82. PMC 3261713. PMID 22068332.
- ^ Wu L, Candille SI, Choi Y, Xie D, Jiang L, Li-Pook-Than J, Tang H, Snyder M (juli 2013). "Variation and genetic control of protein abundance in humans". Nature. 499 (7456): 79–82. Bibcode:2013Natur.499...79W. doi:10.1038/nature12223. PMC 3789121. PMID 23676674.
- ^ Dobson CM (2000). "The nature and significance of protein folding". u Pain RH (ured.). Mechanisms of Protein Folding. Oxford, Oxfordshire: Oxford University Press. str. 1–28. ISBN 978-0-19-963789-8.
- ^ Kozlowski LP (januar 2017). "Proteome-pI: proteome isoelectric point database". Nucleic Acids Research. 45 (D1): D1112–D1116. doi:10.1093/nar/gkw978. PMC 5210655. PMID 27789699.
- ^ Fulton AB, Isaacs WB (april 1991). "Titin, a huge, elastic sarcomeric protein with a probable role in morphogenesis". BioEssays. 13 (4): 157–61. doi:10.1002/bies.950130403. PMID 1859393. S2CID 20237314.
- ^ Bruckdorfer T, Marder O, Albericio F (februar 2004). "From production of peptides in milligram amounts for research to multi-tons quantities for drugs of the future". Current Pharmaceutical Biotechnology. 5 (1): 29–43. doi:10.2174/1389201043489620. PMID 14965208.
- ^ Schwarzer D, Cole PA (decembar 2005). "Protein semisynthesis and expressed protein ligation: chasing a protein's tail". Current Opinion in Chemical Biology. 9 (6): 561–69. doi:10.1016/j.cbpa.2005.09.018. PMID 16226484.
- ^ Kent SB (februar 2009). "Total chemical synthesis of proteins". Chemical Society Reviews. 38 (2): 338–51. doi:10.1039/b700141j. PMID 19169452.
- ^ Fernández A, Scott R (septembar 2003). "Dehydron: a structurally encoded signal for protein interaction". Biophysical Journal. 85 (3): 1914–28. Bibcode:2003BpJ....85.1914F. doi:10.1016/S0006-3495(03)74619-0. PMC 1303363. PMID 12944304.
- ^ Davey NE, Van Roey K, Weatheritt RJ, Toedt G, Uyar B, Altenberg B, Budd A, Diella F, Dinkel H, Gibson TJ (januar 2012). "Attributes of short linear motifs". Molecular BioSystems. 8 (1): 268–81. doi:10.1039/c1mb05231d. PMID 21909575.
- ^ a b Voet D, Voet JG. (2004). Biochemistry Vol 1 3rd ed. Wiley: Hoboken, NJ.
- ^ Sankaranarayanan R, Moras D (2001). "The fidelity of the translation of the genetic code". Acta Biochimica Polonica. 48 (2): 323–35. doi:10.18388/abp.2001_3918. PMID 11732604.
- ^ Bairoch A (januar 2000). "The ENZYME database in 2000" (PDF). Nucleic Acids Research. 28 (1): 304–05. doi:10.1093/nar/28.1.304. PMC 102465. PMID 10592255. Arhivirano s originala (PDF), 1. 6. 2011.
- ^ Radzicka A, Wolfenden R (januar 1995). "A proficient enzyme". Science. 267 (5194): 90–3. Bibcode:1995Sci...267...90R. doi:10.1126/science.7809611. PMID 7809611.
- ^ EBI External Services (20. 1. 2010). "The Catalytic Site Atlas at The European Bioinformatics Institute". Ebi.ac.uk. Arhivirano s originala, 3. 8. 2013. Pristupljeno 16. 1. 2011.
- ^ Pickel B, Schaller A (oktobar 2013). "Dirigent proteins: molecular characteristics and potential biotechnological applications". Applied Microbiology and Biotechnology. 97 (19): 8427–38. doi:10.1007/s00253-013-5167-4. PMID 23989917. S2CID 1896003.
- ^ Rüdiger H, Siebert HC, Solís D, Jiménez-Barbero J, Romero A, von der Lieth CW, Diaz-Mariño T, Gabius HJ (april 2000). "Medicinal chemistry based on the sugar code: fundamentals of lectinology and experimental strategies with lectins as targets". Current Medicinal Chemistry. 7 (4): 389–416. doi:10.2174/0929867003375164. PMID 10702616.
- ^ Mulder NJ (28. 9. 2007). "Protein Family Databases". eLS (jezik: engleski). Chichester, UK: John Wiley & Sons, Ltd. str. a0003058.pub2. doi:10.1002/9780470015902.a0003058.pub2. ISBN 978-0-470-01617-6.
- ^ Sisu C, Pei B, Leng J, Frankish A, Zhang Y, Balasubramanian S, et al. (septembar 2014). "Comparative analysis of pseudogenes across three phyla". Proceedings of the National Academy of Sciences of the United States of America. 111 (37): 13361–6. Bibcode:2014PNAS..11113361S. doi:10.1073/pnas.1407293111. PMC 4169933. PMID 25157146.
- ^ a b Guzmán GI, Sandberg TE, LaCroix RA, Nyerges Á, Papp H, de Raad M, et al. (april 2019). "Enzyme promiscuity shapes adaptation to novel growth substrates". Molecular Systems Biology. 15 (4): e8462. doi:10.15252/msb.20188462. PMC 6452873. PMID 30962359.
- ^ Roohi, Bano K, Kuddus M, Zaheer MR, Zia Q, Khan MF, Ashraf GM, Gupta A, Aliev G (2017). "Microbial Enzymatic Degradation of Biodegradable Plastics". Current Pharmaceutical Biotechnology. 18 (5): 429–440. doi:10.2174/1389201018666170523165742. PMID 28545359.
- ^ Hey J, Posch A, Cohen A, Liu N, Harbers A (2008). "Fractionation of complex protein mixtures by liquid-phase isoelectric focusing". 2D PAGE: Sample Preparation and Fractionation. Methods in Molecular Biology. 424. str. 225–39. doi:10.1007/978-1-60327-064-9_19. ISBN 978-1-58829-722-8. PMID 18369866.
- ^ Terpe K (januar 2003). "Overview of tag protein fusions: from molecular and biochemical fundamentals to commercial systems". Applied Microbiology and Biotechnology. 60 (5): 523–33. doi:10.1007/s00253-002-1158-6. PMID 12536251. S2CID 206934268.
- ^ Stepanenko OV, Verkhusha VV, Kuznetsova IM, Uversky VN, Turoverov KK (august 2008). "Fluorescent proteins as biomarkers and biosensors: throwing color lights on molecular and cellular processes". Current Protein & Peptide Science. 9 (4): 338–69. doi:10.2174/138920308785132668. PMC 2904242. PMID 18691124.
- ^ Yuste R (decembar 2005). "Fluorescence microscopy today". Nature Methods. 2 (12): 902–4. doi:10.1038/nmeth1205-902. PMID 16299474. S2CID 205418407.
- ^ Margolin W (januar 2000). "Green fluorescent protein as a reporter for macromolecular localization in bacterial cells". Methods. 20 (1): 62–72. doi:10.1006/meth.1999.0906. PMID 10610805.
- ^ Walker JH, Wilson K (2000). Principles and Techniques of Practical Biochemistry. Cambridge, UK: Cambridge University Press. str. 287–89. ISBN 978-0-521-65873-7.
- ^ Mayhew TM, Lucocq JM (august 2008). "Developments in cell biology for quantitative immunoelectron microscopy based on thin sections: a review". Histochemistry and Cell Biology. 130 (2): 299–313. doi:10.1007/s00418-008-0451-6. PMC 2491712. PMID 18553098.
- ^ Hohsaka T, Sisido M (decembar 2002). "Incorporation of non-natural amino acids into proteins". Current Opinion in Chemical Biology. 6 (6): 809–15. doi:10.1016/S1367-5931(02)00376-9. PMID 12470735.
- ^ Cedrone, Frédéric; Ménez, André; Quéméneur, Eric (1. 8. 2000). "Tailoring new enzyme functions by rational redesign". Current Opinion in Structural Biology (jezik: engleski). 10 (4): 405–410. doi:10.1016/S0959-440X(00)00106-8. ISSN 0959-440X.
- ^ Görg, Angelika; Weiss, Walter; Dunn, Michael J. (2004-12). "Current two-dimensional electrophoresis technology for proteomics". PROTEOMICS (jezik: engleski). 4 (12): 3665–3685. doi:10.1002/pmic.200401031. Provjerite vrijednost datuma u parametru:
|date=(pomoć) - ^ Conrotto, P.; Souchelnytskyi, S. (2008-09). "Proteomic approaches in biological and medical sciences: principles and applications". Experimental Oncology. 30 (3): 171–180. ISSN 1812-9269. PMID 18806738. Provjerite vrijednost datuma u parametru:
|date=(pomoć) - ^ Koegl, M.; Uetz, P. (22. 1. 2008). "Improving yeast two-hybrid screening systems". Briefings in Functional Genomics and Proteomics (jezik: engleski). 6 (4): 302–312. doi:10.1093/bfgp/elm035. ISSN 1473-9550.
- ^ Plewczyński, Dariusz; Ginalski, Krzysztof (1. 3. 2009). "The interactome: Predicting the protein-protein interactions in cells". Cellular and Molecular Biology Letters (jezik: engleski). 14 (1): 1–22. doi:10.2478/s11658-008-0024-7. ISSN 1689-1392. PMC 6275871. PMID 18839074.CS1 održavanje: format PMC-a (link)
- ^ Zhang, Chao; Kim, Sung-Hou (1. 2. 2003). "Overview of structural genomics: from structure to function". Current Opinion in Chemical Biology (jezik: engleski). 7 (1): 28–32. doi:10.1016/S1367-5931(02)00015-7. ISSN 1367-5931.
- ^ Gonen T, Cheng Y, Sliz P, Hiroaki Y, Fujiyoshi Y, Harrison SC, Walz T (decembar 2005). "Lipid-protein interactions in double-layered two-dimensional AQP0 crystals". Nature. 438 (7068): 633–38. Bibcode:2005Natur.438..633G. doi:10.1038/nature04321. PMC 1350984. PMID 16319884.
- ^ Standley DM, Kinjo AR, Kinoshita K, Nakamura H (juli 2008). "Protein structure databases with new web services for structural biology and biomedical research". Briefings in Bioinformatics. 9 (4): 276–85. doi:10.1093/bib/bbn015. PMID 18430752.
- ^ Walian P, Cross TA, Jap BK (2004). "Structural genomics of membrane proteins". Genome Biology. 5 (4): 215. doi:10.1186/gb-2004-5-4-215. PMC 395774. PMID 15059248.
- ^ Sleator RD (2012). "Prediction of protein functions". Functional Genomics. Methods in Molecular Biology. 815. str. 15–24. doi:10.1007/978-1-61779-424-7_2. ISBN 978-1-61779-423-0. PMID 22130980.
- ^ Zhang Y (juni 2008). "Progress and challenges in protein structure prediction". Current Opinion in Structural Biology. 18 (3): 342–48. doi:10.1016/j.sbi.2008.02.004. PMC 2680823. PMID 18436442.
- ^ Xiang Z (juni 2006). "Advances in homology protein structure modeling". Current Protein & Peptide Science. 7 (3): 217–27. doi:10.2174/138920306777452312. PMC 1839925. PMID 16787261.
- ^ Zhang Y, Skolnick J (januar 2005). "The protein structure prediction problem could be solved using the current PDB library". Proceedings of the National Academy of Sciences of the United States of America. 102 (4): 1029–34. Bibcode:2005PNAS..102.1029Z. doi:10.1073/pnas.0407152101. PMC 545829. PMID 15653774.
- ^ Kuhlman B, Dantas G, Ireton GC, Varani G, Stoddard BL, Baker D (novembar 2003). "Design of a novel globular protein fold with atomic-level accuracy". Science. 302 (5649): 1364–68. Bibcode:2003Sci...302.1364K. doi:10.1126/science.1089427. PMID 14631033. S2CID 1939390.
- ^ Ward JJ, Sodhi JS, McGuffin LJ, Buxton BF, Jones DT (mart 2004). "Prediction and functional analysis of native disorder in proteins from the three kingdoms of life". Journal of Molecular Biology. 337 (3): 635–45. doi:10.1016/j.jmb.2004.02.002. PMID 15019783.
- ^ Tompa P, Fersht A (18. 11. 2009). Structure and Function of Intrinsically Disordered Proteins. CRC Press. ISBN 978-1-4200-7893-0. Arhivirano s originala, 19. 4. 2017. Pristupljeno 19. 10. 2016.
- ^ Ritchie DW (februar 2008). "Recent progress and future directions in protein-protein docking". Current Protein & Peptide Science. 9 (1): 1–15. CiteSeerX 10.1.1.211.4946. doi:10.2174/138920308783565741. PMID 18336319.
- ^ Zagrovic B, Snow CD, Shirts MR, Pande VS (novembar 2002). "Simulation of folding of a small alpha-helical protein in atomistic detail using worldwide-distributed computing". Journal of Molecular Biology. 323 (5): 927–37. CiteSeerX 10.1.1.142.8664. doi:10.1016/S0022-2836(02)00997-X. PMID 12417204.
- ^ Herges T, Wenzel W (januar 2005). "In silico folding of a three helix protein and characterization of its free-energy landscape in an all-atom force field". Physical Review Letters. 94 (1): 018101. arXiv:physics/0310146. Bibcode:2005PhRvL..94a8101H. doi:10.1103/PhysRevLett.94.018101. PMID 15698135. S2CID 1477100.
- ^ Hoffmann M, Wanko M, Strodel P, König PH, Frauenheim T, Schulten K, Thiel W, Tajkhorshid E, Elstner M (august 2006). "Color tuning in rhodopsins: the mechanism for the spectral shift between bacteriorhodopsin and sensory rhodopsin II". Journal of the American Chemical Society. 128 (33): 10808–18. doi:10.1021/ja062082i. PMID 16910676.
- ^ Mendive-Tapia D, Mangaud E, Firmino T, de la Lande A, Desouter-Lecomte M, Meyer HD, Gatti F (2018). "Multidimensional Quantum Mechanical Modeling of Electron Transfer and Electronic Coherence in Plant Cryptochromes: The Role of Initial Bath Conditions". J. Phys. Chem. B. 122 (1): 126–136. doi:10.1021/acs.jpcb.7b10412. PMID 29216421.
- ^ Strümpfer J, Schulten K (2012). "Open Quantum Dynamics Calculations with the Hierarchy Equations of Motion on Parallel Computers". J. Chem. Theory Comput. 8 (8): 2808–2816. doi:10.1021/ct3003833. PMC 3480185. PMID 23105920.
- ^ Scheraga HA, Khalili M, Liwo A (2007). "Protein-folding dynamics: overview of molecular simulation techniques". Annual Review of Physical Chemistry. 58: 57–83. Bibcode:2007ARPC...58...57S. doi:10.1146/annurev.physchem.58.032806.104614. PMID 17034338.
- ^ Muñoz-Huerta, Rafael F.; Guevara-Gonzalez, Ramon G.; Contreras-Medina, Luis M.; Torres-Pacheco, Irineo; Prado-Olivarez, Juan; Ocampo-Velazquez, Rosalia V. (Aug 16, 2013). "A Review of Methods for Sensing the Nitrogen Status in Plants: Advantages, Disadvantages and Recent Advances". Sensors (Basel, Switzerland). 13 (8): 10823–10843. Bibcode:2013Senso..1310823M. doi:10.3390/s130810823. PMC 3812630. PMID 23959242.
- ^ Martin, P D; Malley, D F; Manning, G.; Fuller, L. (Nov 1, 2002). "Determination of soil organic carbon and nitrogen at the field level using near-infrared spectroscopy". Canadian Journal of Soil Science. 82 (4): 413–422. doi:10.4141/S01-054 – preko DOI.org (Crossref).
- ^ Brosnan JT (juni 2003). "Interorgan amino acid transport and its regulation". The Journal of Nutrition. 133 (6 Suppl 1): 2068S–72S. doi:10.1093/jn/133.6.2068S. PMID 12771367.
- ^ Watson TD (1998). "Diet and skin disease in dogs and cats". The Journal of Nutrition. 128 (12 Suppl): 2783S–89S. doi:10.1093/jn/128.12.2783S. PMID 9868266.
- ^ a b Case LP, Daristotle L, Hayek MG, Raasch MF (2010). Canine and Feline Nutrition-E-Book: A Resource for Companion Animal Professionals. Elsevier Health Sciences.
Dopunska literatura
[uredi | uredi izvor]- Udžbenici
- Branden C, Tooze J (1999). Introduction to Protein Structure. New York: Garland Pub. ISBN 978-0-8153-2305-1.
- Murray RF, Harper HW, Granner DK, Mayes PA, Rodwell VW (2006). Harper's Illustrated Biochemistry. New York: Lange Medical Books/McGraw-Hill. ISBN 978-0-07-146197-9.
- Van Holde KE, Mathews CK (1996). Biochemistry. Menlo Park, California: Benjamin/Cummings Pub. Co., Inc. ISBN 978-0-8053-3931-4.
Vanjski linkovi
[uredi | uredi izvor]Baze podataka i projekti
[uredi | uredi izvor]- NCBI Entrez Protein database
- NCBI Protein Structure database
- Human Protein Reference Database
- Human Proteinpedia
- Folding@Home (Stanford University) Arhivirano 8. 9. 2012. na Wayback Machine
- Protein Databank in Europe (see also PDBeQuips[mrtav link], short articles and tutorials on interesting PDB structures)
- Research Collaboratory for Structural Bioinformatics (see also Molecule of the Month Arhivirano 24. 7. 2020. na Wayback Machine, presenting short accounts on selected proteins from the PDB)
- Proteopedia – Life in 3D: rotatable, zoomable 3D model with wiki annotations for every known protein molecular structure.
- UniProt the Universal Protein Resource
Tutorijali i obrazovne stranice
[uredi | uredi izvor]- "An Introduction to Proteins" from HOPES (Huntington's Disease Outreach Project for Education at Stanford)
- Proteins: Biogenesis to Degradation – The Virtual Library of Biochemistry and Cell Biology

