리보솜 RNA
Ribosomal RNA| rRNA | |
|---|---|
 다양한 종류의 rRNA | |
| 식별자 | |
| 기타 데이터 | |
| RNA형 | 진; rRNA |
| PDB 구조 | PDBe |
리보솜 리보핵산(rRNA)은 모든 세포에 필수적인 리보솜의 1차 성분인 비코드 RNA의 일종이다.리보솜 RNA는 리보솜 DNA(rDNA)에서 전사된 후 리보솜 단백질에 결합되어 작고 큰 리보솜 서브유닛을 형성한다.rRNA는 전달 RNA(tRNA)와 메신저 RNA(mRNA)가 [1]후자를 처리하고 번역하도록 강요하는 리보솜의 물리적, 기계적 요인이다.리보솜 RNA는 대부분의 세포에서 발견되는 RNA의 지배적인 형태이다; 그것은 단백질로 변환되지 않음에도 불구하고 세포 RNA의 약 80%를 구성한다.리보솜은 질량 기준 약 60%의 rRNA와 40%의 리보솜 단백질로 구성되어 있다.
구조.
rRNA 배열의 1차 구조는 유기체에 따라 다를 수 있지만, 이러한 배열 내의 염기쌍은 일반적으로 스템 루프 구성을 형성한다.이러한 rRNA 스템루프의 길이와 위치는 여러 [2]종에서 유사한 3차원 rRNA 구조를 만들 수 있게 한다.이러한 구성 때문에, rRNA는 리보솜 단백질과 긴밀하고 특정한 상호작용을 형성하여 리보솜 서브유닛을 형성할 수 있다.이러한 리보솜 단백질은 염기성 잔류물(산성 잔류물과는 대조적으로)과 방향족 잔류물(즉, 페닐알라닌, 티로신 및 트립토판)을 포함하고 있어 쌓기 상호작용과 같은 관련 RNA 영역과 화학적 상호작용을 형성할 수 있다.리보솜 단백질은 또한 염기성 잔류물(리신 및 아르기닌)로 구성된 결합 부위와 rRNA의 당-인산 골격에 가교할 수 있다.모든 리보솜 단백질(rRNA에 결합하는 특정 배열 포함)이 확인되었다.이러한 상호작용은 작고 큰 리보솜 서브유닛의 연관성과 함께 단백질을 [3]합성할 수 있는 기능성 리보솜을 생성한다.
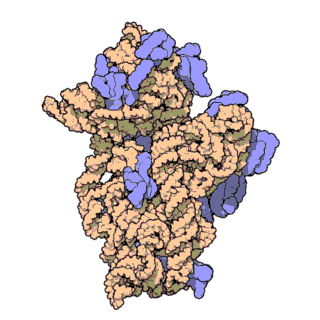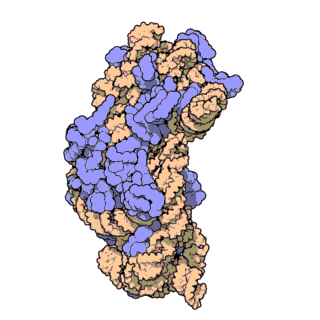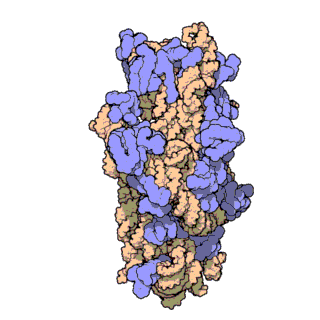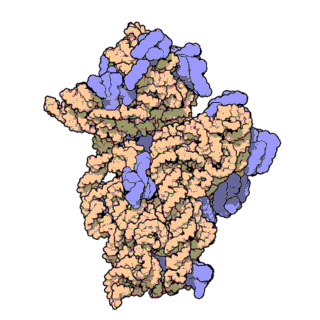
리보솜 RNA는 큰 서브유닛(LSU)과 작은 서브유닛(SSU)의 두 가지 주요 리보솜 서브유닛으로 구성됩니다.각 유형 중 하나가 함께 모여 기능하는 리보솜을 형성합니다.서브유닛은 때때로 크기-침강 측정값("S" 접미사가 있는 숫자)으로 참조됩니다.원핵생물에서 LSU와 SSU는 각각 50S와 30S 서브유닛으로 불린다.진핵생물에서, 그것들은 조금 더 크다; 진핵생물의 LSU와 SSU는 각각 60S와 40S 서브 유닛이라고 불린다.
박테리아와 같은 원핵생물의 리보솜에서 SSU는 단일 작은 rRNA 분자(~1500 뉴클레오티드)를 포함하는 반면 LSU는 단일 작은 rRNA 분자 및 단일 큰 rRNA 분자(~3000 뉴클레오티드)를 포함합니다.이것들은 50개까지의 리보솜 단백질과 결합되어 리보솜 서브유닛을 형성한다.원핵생물 리보솜에서 발견되는 rRNA에는 LSU의 23S 및 5S rRNA와 SSU의 16S rRNA의 세 가지 유형이 있습니다.
사람과 같은 진핵생물의 리보솜에서 SSU는 단일 작은 rRNA(~1800 뉴클레오티드)를 포함하는 반면 LSU는 두 개의 작은 rRNA와 한 분자의 큰 rRNA(~5000 뉴클레오티드)를 포함합니다.진핵생물 [4]rRNA는 원핵생물에 비해 더 크고 더 많은 다형성 리보솜 단위를 형성하기 위해 상호작용하는 70개 이상의 리보솜 단백질을 가지고 있다.진핵생물에는 LSU에 3종, SSU에 [5]1종 등 4종류의 rRNA가 있으며, 효모는 진핵생물 rRNA의 행동과 과정을 관찰하는 전통적인 모델로서 연구 다양화의 부족을 초래하고 있다.기술적 진보(특히 Cryo-EM 분야)가 다른 진핵생물에서 [6]리보솜 거동에 대한 예비 조사를 가능하게 한 것은 지난 10년 이내이다.효모에서 LSU는 5S, 5.8S 및 28S rRNA를 포함한다.결합된 5.8S와 28S는 진핵생물에서만 발생하는 것으로 생각되었던 리보솜 표면에 국소화된 원핵생물 23S rRNA 서브타입 마이너스 확장 세그먼트(ES)와 크기와 기능이 대략 동일하다.하지만 최근, 진핵아에 가장 가까운 고고학적 근연으로 여겨지는 아스가르드 필라, 즉 로키아르카이오타와 헤임달라카이오타는 그들의 23S rRNA에 [7]두 개의 초대형 ES를 가지고 있는 것으로 보고되었다.마찬가지로 5S rRNA는 할로콘성 고균의 리보솜에 [8][9]108μ뉴클레오티드 삽입을 포함한다.
진핵생물 SSU는 ES도 포함하는 18S rRNA 서브유닛을 포함한다.SSU ES는 일반적으로 LSU ES보다 작습니다.
SSU 및 LSU rRNA 배열은 고대 생명체의 [10]모든 알려진 형태에서 발견되고 수평적 유전자 전달에 저항하기 때문에 유기체 간의 진화 관계에 대한 연구에 널리 사용됩니다. rRNA 배열은 [11]리보솜의 기능에서 중요한 역할을 하기 때문에 시간이 지남에 따라 보존(변화되지 않음)됩니다.16s rRNA에서 파생된 계통발생 정보는 현재 뉴클레오티드 [12]유사도를 계산함으로써 유사한 원핵종 간의 주요 묘사 방법으로 사용되고 있다.생명의 정석나무는 번역 시스템의 계통이다.
LSU rRNA 아형은 리보솜 단백질이 리보솜의 촉매 부위와 결합할 수 없기 때문에 리보자임이라고 불렸다(특히 펩티딜전달효소 중심, PTC).[13]
SSU rRNA 서브타입은 디코딩센터(DC)[14]에서 mRNA를 디코딩합니다.리보솜 단백질은 DC로 들어갈 수 없다.
rRNA의 구조는 다른 [15]mRNA의 번역 중에 리보솜에 대한 tRNA 결합에 영향을 미치도록 급격하게 변할 수 있다.16s rRNA에서, 이것은 rRNA의 특정 뉴클레오티드가 하나의 뉴클레오티드 또는 다른 뉴클레오티드 사이에 번갈아 염기쌍을 형성하여 rRNA의 구조를 바꾸는 "스위치"를 형성할 때 발생하는 것으로 생각된다.이 프로세스는 LSU 및 SSU의 구조에 영향을 줄 수 있습니다.이는 rRNA 구조 내의 이 컨포메이션스위치가 tRNA 선택 시 코돈과 안티코돈을 일치시키는 능력 및 mRNA [16]디코딩에 있어서 리보솜 전체에 영향을 미친다는 것을 나타냅니다.
어셈블리
리보솜 RNA의 리보솜으로의 통합 및 조립은 리보솜 단백질과의 접힘, 수정, 가공 및 조립으로 시작하여 두 개의 리보솜 서브 유닛, LSU와 SSU를 형성합니다. 원핵생물에서는 막 결합 오르가넬의 부족으로 인해 세포질에서 RNA 혼입이 발생합니다.그러나 진핵생물에서 이 과정은 주로 핵에서 일어나며 사전 RNA의 합성에 의해 시작된다.이를 위해서는 세 가지 RNA 중합효소가 모두 존재해야 합니다.사실, RNA 중합효소 I에 의한 사전 RNA 전사는 세포의 전체 RNA [17]전사의 약 60%를 차지한다.이것은 리보솜 단백질과 조립될 수 있도록 사전 RNA의 접힘이 뒤따른다.이러한 접힘은 엔도핵산 및 엑소핵산 분해효소, RNA 헬리케이스, GTPase 및 ATPase에 의해 촉매된다.rRNA는 이후 외부 및 내부 전사 스페이서를 [18]제거하기 위해 내핵 및 외핵 분해 처리를 거친다.그런 다음 프리 RNA는 리보솜 조립 인자와 리보솜 단백질이 프리 RNA와 결합하여 프리 리보솜 입자를 형성하기 전에 메틸화 또는 의사우리디닐화와 같은 변형을 겪는다.더 성숙 단계를 거쳐 핵에서 세포질로 나갈 때, 이 입자들은 리보솜을 [18]형성하기 위해 결합합니다.rRNA의 1차 구조 내에서 발견되는 염기성 및 방향족 잔류물은 rRNA의 골격과 리보솜 단위의 다른 성분들 사이에 가교 효과를 발생시키면서 리보솜 단백질에 대한 바람직한 적층 상호작용과 흡인을 가능하게 한다.이러한 프로세스의 시작 및 시작 부분에 대한 자세한 내용은 "생합성" 섹션에서 확인할 수 있습니다.
기능.
서로 다른 종들 사이에서 rRNA에서 보편적으로 보존된 2차 구조 요소들은 이러한 배열들이 가장 오래된 것 중 일부라는 것을 보여준다.이들은 mRNA 번역의 촉매 부위를 형성하는 데 중요한 역할을 합니다.mRNA의 번역 중에 rRNA는 mRNA와 tRNA를 모두 결합하여 mRNA의 코돈 배열을 아미노산으로 변환하는 과정을 용이하게 한다.rRNA는 tRNA가 SSU와 LSU 사이에 끼었을 때 단백질 합성의 촉매를 개시한다. SSU의 mRNA는 항문제와 상호작용한다.LSU에서 tRNA의 아미노산 수용체 스템은 LSU rRNA와 상호작용한다.리보솜은 에스테르-아미드 교환을 촉매하여 초기 펩타이드의 C 말단을 tRNA에서 아미노산의 아민으로 전달합니다.이러한 과정은 이러한 분자들이 rRNA 줄기 고리에 의해 형성되어 결합할 수 있는 리보솜 내의 부위 때문에 발생할 수 있습니다.리보솜에는 A, P 및 E 사이트라고 불리는 3개의 결합 사이트가 있습니다.
- 일반적으로 A(아미노아실) 부위는 아미노아실-tRNA(3' 말단에서 아미노산으로 에스테르화된 tRNA)를 포함한다.
- P(펩티딜) 부위에는 초기 펩타이드에 에스테르화된 tRNA가 포함되어 있습니다.A 부위 tRNA의 유리 아미노기(NH2)는 P 부위 tRNA의 에스테르 결합을 공격하여 초기 펩타이드를 A 부위 아미노산으로 전달시킨다.이 반응은 펩티딜전달효소[13] 중심에서 일어난다.
- E(출구) 부위에는 방출된 tRNA가 포함되어 있으며, (아미노산 또는 신생 펩타이드 없음) 3' 말단이 자유롭다.
단일 mRNA는 여러 리보솜에 의해 동시에 번역될 수 있다.이것은 폴리썸이라고 불립니다.
원핵생물에서는 mRNA의 번역에 있어 rRNA의 중요성을 확인하기 위해 많은 연구가 이루어졌다.예를 들어 A 부위는 주로 16S rRNA로 구성되며, 이 부위에서 tRNA와 상호작용하는 다양한 단백질 원소를 제외하고 리보솜 구조를 변경하지 않고 이들 단백질을 제거하면 해당 부위는 정상적으로 기능할 것이라는 가설을 가지고 있다.P부위에서는 결정구조 관찰을 통해 16s rRNA의 3' 말단이 mRNA 분자처럼 접힐 수 있는 것으로 나타났다.이는 서브유닛을 안정시키는 분자간 상호작용을 일으킨다.마찬가지로, A 부위와 마찬가지로, P 부위는 주로 단백질이 거의 없는 rRNA를 포함한다.예를 들어 펩티딜전달효소 중심은 23S rRNA 서브유닛의 [13]뉴클레오티드에 의해 형성된다.실제로 펩티딜전달효소 중심은 단백질을 포함하지 않으며, 전적으로 rRNA의 존재에 의해 개시된다는 연구결과가 있다.A와 P 부위와 달리 E 부위는 더 많은 단백질을 포함하고 있다.단백질은 A와 P 부위의 기능에 필수적이지 않기 때문에, E 부위의 분자 조성은 아마도 나중에 진화했다는 것을 보여준다.원시 리보솜에서는 tRNA가 P 부위에서 빠져나갔을 가능성이 있다.또한 E-사이트 tRNA가 16S [19]및 23S rRNA 서브유닛과 결합하는 것으로 나타났다.
서브유닛 및 관련 리보솜 RNA
원핵과 진핵 리보솜은 둘 다 큰 단위와 작은 단위 두 가지로 나눌 수 있다.각각의 rRNA에 대해 아래 표에 사용된 예시적인 종은 대장균(원핵생물)과 인간(유카리오테)이다."nt"는 뉴클레오티드에서 rRNA 유형의 길이를 나타내며, "S"("16S")는 스베드베르크 단위를 나타낸다.
| 유형 | 크기 | 대형 서브유닛(LSU rRNA) | 소형 서브유닛(SSU rRNA) |
| 원핵생물 | 70S | 50S(5S: 120nt, 23S: 2906nt) | 30S(16S: 1542nt) |
| 진핵생물 | 80S | 60S(5S: 121nt,[20] 5.8S: 156nt,[21] 28S: 5070nt[22]) | 40S(18S: 1869[23] nt) |
서브유닛(또는 rRNA)의 S단위는 질량이 아닌 침강속도를 나타내기 때문에 단순히 추가할 수 없다.각 서브유닛의 침강속도는 그 모양과 질량의 영향을 받는다.nt 단위는 선형 rRNA 중합체의 정수 단위 수(예를 들어 인간 rRNA = 7216nt)를 나타내므로 추가될 수 있다.
rRNA를 코드하는 유전자 클러스터는 일반적으로 "리보솜 DNA" 또는 rDNA라고 불린다.
원핵생물에서
원핵생물에서 작은 30S 리보솜 서브유닛은 16S 리보솜 RNA를 포함한다.큰 50S 리보솜 서브유닛은 두 개의 rRNA 종(5S 및 23S 리보솜 RNA)을 포함한다.따라서 박테리아와 고세균 모두에서 16S, 23S,[24] 5S의 세 가지 rRNA 유형을 모두 코드하는 하나의 rRNA 유전자가 존재한다고 추론할 수 있다.
박테리아 16S 리보솜 RNA, 23S 리보솜 RNA 및 5S rRNA 유전자는 일반적으로 공전사 오퍼론으로 구성된다.본 섹션의 이미지와 같이, 16S와 23S의 rRNA [25]유전자 사이에는 내부 전사 스페이서가 존재한다.게놈에 하나 이상의 오퍼론의 복사본이 분산되어 있을 수 있습니다(예를 들어 대장균은 7개입니다).일반적으로 박테리아에는 [24]1개에서 15개의 복사본이 있습니다.
고세균은 단일 rRNA 유전자 오퍼론 또는 최대 4개의 동일한 오퍼론 [24]복사본을 포함합니다.
16S 리보솜 RNA의 3' 말단(리보솜 내)은 샤인-달가르노 배열이라고 불리는 mRNA의 5' 말단에서 배열을 인식한다.
진핵생물에서
대조적으로, 진핵 생물은 일반적으로 연속적으로 배열된 rRNA 유전자의 많은 복사본을 가지고 있다.사람의 경우, 염색체 13(RNR1), 14(RNR2), 15(RNR3), 21(RNR4) 및 22(RNR5)에 위치한 5개의 클러스터에 약 300–400번의 반복이 존재한다.이배체 인간은 10개의 게놈 rDNA 클러스터를 가지고 있으며, 이는 총 인간 [26]게놈의 0.5% 미만을 차지한다.
이전에 반복 rDNA 시퀀스가 동일하고 자연 복제 오류와 포인트 돌연변이를 설명하기 위한 중복 또는 실패의 역할을 하는 것으로 받아들여졌다.그러나 다중 염색체에 걸친 인간의 rDNA(그리고 그 후 rRNA)의 염기서열 변화는 인간 개인 내 및 개인 간에 관찰되었다.이러한 변이의 대부분은 회문 배열과 [27]복제로 인한 잠재적 오류입니다.특정 변종들은 [28]또한 마우스에서 조직 고유의 방식으로 발현된다.
포유류의 세포는 2개의 미토콘드리아(12S, 16S) rRNA 분자와 4종의 세포질 rRNA(28S, 5.8S, 18S, 5S 서브유닛)를 가지고 있다.28S, 5.8S 및 18S의 rRNA는 내부에서 변환된2개의 스페이서로 구분된1개의 Transcription Unit(45S)에 의해 부호화 됩니다.첫 번째 스페이서는 박테리아와 고세균에서 발견되는 스페이서와 일치하고, 다른 스페이서는 원핵생물에서 [25]23S rRNA였던 것에 대한 삽입입니다.45S rDNA는 염색체 13, 14, 15, 21 및 22에 5개의 클러스터(각각 30-40회 반복)로 구성된다.이것들은 RNA 중합효소 I에 의해 전사된다.5S 서브유닛의 DNA는 염색체 1q41-42에서 가장 큰 배열(~200–300개의 진짜 5S 유전자와 많은 분산된 유사 유전자)로 발생한다. 5S rRNA는 RNA 중합효소 III에 의해 전사된다.대부분의 진핵생물에서 18S rRNA는 작은 리보솜 서브유닛에 있고, 큰 서브유닛은 3개의 rRNA 종(포유동물에서 5S, 5.8S, 28S, 식물에서 25S, rRNA)을 포함한다.
소형 서브유닛 리보솜 RNA(SSU rRNA)의 3차 구조는 X선 결정학에 [29]의해 분해되었다.SSU rRNA의 세컨더리 구조에는 4개의 도메인이 있습니다.즉, 5', central, 3' 메이저 및 3' 마이너 도메인이 포함됩니다.5' 도메인(500~800 뉴클레오티드)의 2차 구조 모델이 표시된다.
생합성
진핵생물에서
소자의 구성 요소로서 rRNA의 생산은 궁극적으로 리보솜 합성의 속도 제한 단계이다.핵에서 rRNA는 그것을 코드하는 특수 유전자(rDNA)를 사용하여 RNA 중합효소 I에 의해 합성되며,[30] 이는 게놈 전체에서 반복적으로 발견된다.18S, 28S 및 5.8S rRNA를 코드하는 유전자는 핵산 조직체 영역에 위치하며 RNA 중합효소 I에 의해 큰 전구체 rRNA(pre-rNA) 분자로 변환된다.이러한 사전 RNA 분자는 외부 및 내부 스페이서 배열에 의해 분리된 후 메틸화되며, 이는 나중에 조립 및 [31][32][33]접기에 중요한 요소입니다.개별 분자로 분리 및 방출된 후, 조립 단백질은 각각의 벗은 rRNA 가닥에 결합하고 필요에 따라 공동 조립 및 더 많은 접힘 단백질의 점진적인 추가를 사용하여 기능적 형태로 접힙니다.접힌 단백질이 어떻게 rRNA에 결합하고 어떻게 정확하게 접히는지에 대한 정확한 세부 사항은 알려지지 않았다.[34]그런 다음, 단백질 복합체와 함께 snoRNA(작은 핵소형 RNA)에 의해 유도되는 핵외 및 핵내 분열을 포함한 반응에 의해 rRNA 복합체는 더욱 처리된다.이들 복합체가 결합단위를 형성하기 위해 압축됨에 따라 rRNA와 주변 리보솜 단백질 간의 상호작용은 안정성을 제공하고 결합부위를 [35]보호하기 위해 조립 전체에서 지속적으로 리모델링된다.이 프로세스를 rRNA 라이프 사이클의 "성숙" 단계라고 한다.rRNA의 성숙기에 발생하는 변형은 tRNA와 mRNA의 [36]번역접근에 대한 물리적 조절을 제공함으로써 유전자 발현 제어에 직접 기여하는 것으로 밝혀졌으며, 일부 연구는 리보솜 [37][38]안정성을 유지하기 위해 다양한 rRNA 유형의 광범위한 메틸화도 필요하다는 것을 발견했다.
5S rRNA에 대한 유전자는 뉴클레오스 내부에 위치하며 RNA 중합효소 [39]III에 의해 5S 이전 rRNA로 변환된다.5S 이전 rRNA는 28S 및 5.8S rRNA와 함께 처리 및 조립하기 위해 핵으로 들어가 LSU를 형성하며, 18S rRNA는 수많은 리보솜 단백질과 결합하여 SSU를 형성한다.두 서브유닛이 조립되면 개별적으로 세포질로 내보내져 80S 유닛을 형성하고 mRNA의 [40][41]변환을 시작합니다.
리보솜 RNA는 비코딩이고 어떤 종류의 단백질로도 변환되지 않는다: rRNA는 rDNA로부터만 전사되고 리보솜의 구조적 구성 요소로 사용하기 위해 성숙된다.전사된 rRNA는 리보솜 단백질에 결합되어 리보솜의 서브유닛을 형성하며 mRNA와 tRNA를 리보솜을 통해 밀어내고 그것들을 [1]처리하고 번역하는 물리적 구조로서 작용한다.
진핵생물조절
rRNA 합성은 다양한 과정과 상호작용에 의해 항상성을 유지하기 위해 상향 조절 및 하향 조절된다.
- 효소 AKT는 RNA 중합효소 I이 AKT [42]의존성이기 때문에 간접적으로 rRNA 합성을 촉진한다.
- 앤지제닌(ANG)과 같은 특정 혈관 발생 리보핵산가수분해효소는 핵에 전이되어 축적될 수 있다.ANG의 농도가 너무 높아지면, 일부 연구는 ANG가 rDNA의 프로모터 영역에 결합하고 불필요하게 rRNA 전사를 증가시킬 수 있다는 것을 발견했다.이것은 핵에 해를 끼칠 수 있고 심지어 검사되지 않은 전사와 [43]암을 초래할 수 있다.
- 세포 포도당 제한 기간 동안, AMP 활성화 단백질 키나제(AMPK)는 에너지를 소비하지만 필수적이지 않은 대사 과정을 방해한다.그 결과 전사개시를 [44]방해함으로써 rRNA 합성을 다운조절하기 위해 RNA 중합효소 I(Ser-635 부위)를 인산화시킬 수 있다.
- 리보솜 복호화 중심에서 둘 이상의 슈도우리딘 또는 29-O-메틸화 영역을 손상 또는 제거함으로써 [45]신규 아미노산 혼입 속도를 감소시킴으로써 rRNA 전사 속도를 유의하게 감소시킨다.
- 헤테로크로마틴의 형성은 rRNA 전사를 소음화하는 데 필수적이며, rRNA 전사가 없으면 리보솜 RNA가 억제되지 않고 합성되어 [46]유기체의 수명을 크게 감소시킨다.
원핵생물에서
진핵생물들과 유사하게, rRNA의 생산은 리보솜의 원핵생물 합성의 속도 제한 단계이다.대장균에서 rRNA는 7개의 서로 다른 rrn 오퍼론 내에서 발견된 2개의 프로모터 P1과 P2에서 전사되는 것으로 확인되었다.P1 프로모터는 중간에서 높은 세균 성장률 동안 rRNA 합성을 조절하는 역할을 합니다.이 프로모터의 전사 활성은 성장률과 정비례하기 때문에 주로 rRNA 조절에 관여한다.rRNA 농도의 증가는 리보솜 합성에 대한 부정적인 피드백 메커니즘으로 작용한다.rrn P1 프로모터의 효율적인 문자 변환을 위해서는 높은 NTP 집중이 필요한 것으로 판명되었습니다.이들은 RNA 중합효소 및 촉진제와 안정화 복합체를 형성하는 것으로 생각된다.특히 박테리아에서 높은 NTP 농도와 rRNA 합성의 증가는 리보솜과 단백질 합성이 성장 속도에 의존하는 이유에 대한 분자적 설명을 제공한다.성장률이 낮을수록 rRNA/리보솜 합성 속도가 낮아지고, 성장률이 높을수록 rRNA/리보솜 합성 속도가 높아진다.이것은 세포가 에너지를 절약하거나 필요와 사용 가능한 [47][48][49]자원에 따라 신진대사 활동을 증가시킬 수 있게 해준다.
원핵세포에서 각 rRNA 유전자 또는 오퍼론은 전사된 스페이서와 함께 16S, 23S, 5S rRNA 및 tRNA 서열을 포함하는 단일 RNA 전구체로 전사된다.그런 다음 전사가 완료되기 전에 RNA 처리가 시작됩니다.처리 반응 중에, rRNA와 tRNA는 분리된 [50]분자로 방출된다.
원핵생물 조절
원핵생물의 세포 생리학에서 rRNA가 하는 중요한 역할 때문에, rRNA 조절 메커니즘에는 많은 중복이 있다.전사 수준에서는 세포의 항상성 유지를 촉진하는 rRNA 전사에는 다음과 같은 양의 효과와 음의 효과가 모두 있습니다.
- rrn P1 프로모터의 상류 UP 원소는 RNA 중합효소 서브유닛을 결합할 수 있어 rRNA 전사를 촉진할 수 있다.
- FIS와 같은 전사 인자는 프로모터의 업스트림에 결합하고 전사를 촉진하는 RNA 중합효소와의 상호작용을 한다.
- 항종단 인자는 rrn P2 프로모터의 하류에 결합하여 조기 전사 종단을 방지한다.
- 따라서 아미노산 가용성이 낮을 때 P1 [47]및 P2 프로모터로부터의 전사를 억제할 수 있다.
열화
리보솜 RNA는 다른 일반적인 유형의 RNA에 비해 상당히 안정적이며 건강한 세포 환경에서 장기간 지속됩니다.일단 기능 단위로 조립되면, 리보솜 내의 리보솜 RNA는 세포 수명 주기의 정지 단계에서 [51]수 시간 동안 안정적입니다.열화는 리보솜의 "stalling"을 통해 유발될 수 있습니다. 리보솜은 결함이 있는 mRNA를 인식하거나 리보솜에 의한 변환을 중지시키는 다른 처리상의 어려움을 만났을 때 발생합니다.일단 리보솜이 멈추면,[52] 리보솜의 특수 경로가 시작되어 전체 복합체를 분해 대상으로 삼는다.
진핵생물에서
다른 단백질 또는 RNA와 마찬가지로, rRNA 생산은 오류가 발생하기 쉽고, 결과적으로 비기능적 rRNA 생산으로 이어집니다.이를 교정하기 위해 세포는 비기능적 rRNA 붕괴([53]NRD) 경로를 통해 rRNA의 분해를 허용한다.이 주제에 대한 연구의 대부분은 진핵 세포, 특히 사카로미세스 세레비시아이 효모에 대해 수행되었다.현재, 세포들이 어떻게 진핵 생물의 유비쿼티화와 분해를 위해 기능적으로 결함이 있는 리보솜을 목표로 삼을 수 있는지에 대한 기본적인 이해만 가능하다.[54]
- 40S 서브유닛의 NRD 경로는 60S 서브유닛의 NRD 경로와는 독립적이거나 분리될 수 있다.특정 유전자가 특정 사전 RNA의 분해에 영향을 미칠 수 있었지만 다른 [55]유전자는 영향을 미치지 않는 것으로 관찰되었다.
- Mms1p 및 Rtt101p와 같은 수많은 단백질이 NRD 경로에 관여하고 있으며, 이들은 분해를 위해 리보솜을 목표로 하기 위해 함께 복합되는 것으로 여겨진다.Mms1p와 Rtt101p는 결합하는 것으로 확인되며, Rtt101p는 유비퀴틴 E3 리가아제 착체를 모집하여 비기능성 리보솜이 [56]분해되기 전에 유비퀴틴화될 수 있도록 한다.
- 진핵세포의 성장률은 비기능적 rRNA의 축적에 큰 영향을 받지 않는 것으로 보인다.
원핵생물에서
비록 진핵생물에 비해 원핵생물의 리보솜 RNA 분해에 대한 연구는 훨씬 적지만, 박테리아가 진핵생물의 NRD와 비교하여 유사한 분해 체계를 따르는지에 대해서는 여전히 관심이 있었다.원핵생물을 위해 수행된 연구의 대부분은 대장균에 대해 수행되었다.진핵생물 및 원핵생물 rRNA 분해 사이에 많은 차이점이 발견되어 연구자들이 두 가지가 다른 경로를 [57]통해 분해된다고 믿게 되었다.
- 진핵생물에서 rRNA 분해를 촉발할 수 있었던 rRNA의 특정 돌연변이는 원핵생물에서는 그렇게 할 수 없었다.
- 23S rRNA의 점 돌연변이는 한 서브유닛의 돌연변이가 해당 서브유닛의 저하만 야기하는 진핵생물과 비교하여 23S 및 16S rRNA를 모두 저하시킨다.
- 연구자들은 23S rRNA에서 전체 나선 구조(H69)를 제거해도 분해가 유발되지 않는다는 것을 발견했다.이것은 H69가 돌연변이된 rRNA를 인식하고 분해하는 데 엔도핵산 효소에 매우 중요하다고 믿게 했다.
시퀀스 보존 및 안정성
모든 유기체에 걸친 rRNA의 보편적이고 변함없는 특성으로 인해, 유기체의 파괴 없이 유전자 이동, 돌연변이, 그리고 변화에 대한 rRNA의 저항성에 대한 연구는 인기 있는 관심 분야가 되었다.리보솜 RNA 유전자는 변형과 침입에 내성이 있는 것으로 밝혀졌다.rRNA 배열이 변경되면 세포는 손상되어 정상적인 [58]기능을 빠르게 정지하는 것으로 밝혀졌다.이러한 rRNA의 주요 특성은 유전자 데이터베이스 프로젝트(SILVA 또는[60] SINA와 같은[59] 포괄적인 온라인 자원)에서 특히 중요해졌다. 이 프로젝트에서는 서로 다른 생물학적 영역에서 리보솜 RNA 배열이 "분류학적 할당, 계통발생학적 분석 및 미생물 [59]다양성의 조사"를 크게 용이하게 한다.
복원력의 예:
- 16S rRNA 단위의 많은 부분에 크고 무의미한 RNA 조각이 추가되어도 리보솜 단위 [61]전체의 기능은 눈에 띄게 변화하지 않는다.
- 비부호화RD7 RNA는 rRNA의 처리를 변화시켜 카르본산에 의한 분해에 저항하는 분자를 만드는 능력을 가진다.이것은 (ATP를 생산하는 데 필요한 기질 인산화 때문에) 산 축적이 세포 내 기능에 [62]독성이 될 수 있을 때 활성 성장 동안 rRNA 농도를 유지하는 데 중요한 메커니즘이다.
- 16S rRNA를 따라 시스 분해가 가능한 해머헤드 리보자임을 삽입하면 기능이 크게 저해되고 [61]안정성이 저하됩니다.
- 대부분의 세포 기능이 저산소 환경에 대한 짧은 노출 후 크게 저하되는 반면, rRNA는 저산소증이 지속된 6일 후에도 분해되지 않고 분해된다.이러한 긴 시간이 지난 후에야 rRNA 중간체([63]최종적으로 열화가 발생하는 것을 나타낸다)가 나타나기 시작한다.
중요성
리보솜 RNA의 특성은 진화, 즉 분류학 및 의학에서 중요하다.
- rRNA는 모든 [41]세포에 존재하는 몇 안 되는 유전자 생성물 중 하나이다.이러한 이유로, rRNA(rDNA)를 코드하는 유전자는 유기체의 분류학적 그룹을 식별하고, 관련된 그룹을 계산하고, 종의 [64]발산 속도를 추정하기 위해 배열된다.그 결과 수천 개의 rRNA 시퀀스가 알려져 RDP-II[65] 및 [66]SILVA와 같은 특수한 데이터베이스에 저장됩니다.
- rRNA의 변화는 결핵균(결핵을 일으키는 박테리아)과 같은 특정 질병을 일으키는 박테리아가 극도의 약물 [67]내성을 갖도록 하는 것이다.유사한 문제로 인해, 이것은 애완동물의 세균 감염을 다루는 주요 방법이 박테리아 리보솜의 펩티딜 전달효소 센터(PTC)를 공격하는 약물의 투여인 수의학에서 널리 알려진 문제가 되었다.23S rRNA의 돌연변이는 PTC를 완전히 [68]우회하기 위해 알려지지 않은 방식으로 함께 작용하기 때문에 이러한 약물에 대한 완벽한 내성을 만들어냈다.
- rRNA는 클로람페니콜, 에리트로마이신, 카스가마이신, 마이크로코신, 파로마이신, 리신, 알파사르신, 스펙티노마이신, 스트렙토마이신, 티오스트렙톤과 같은 임상적으로 관련된 수많은 항생제의 대상이다.
- rRNA는 인간의 miR-663과 생쥐의 miR-712와 같은 종 고유의 마이크로RNA의 기원으로 나타났다.이러한 특정 miRNA는 rRNA의 [69]내부 전사 스페이서에서 유래합니다.
인간 유전자
- 45S: RNR1, RNR2, RNR3, RNR4, RNR5, (비클러스터형) RNA18SN1, RNA18SN2, RNA18SN3, RNA18SN4, RNA18SN5, RNA28SN1, 28RNASN2, RNA28SN3, RNA28SN4, RNA28SN5, RNA45SN1, RNA45SN2, RNA45SN3, RNA45SN4, RNA45SN5, RNA5-8SN1, RNA5-8SN2, RNA5-8SN3, RNA5-8SN4, RNA5-8SN5
- 5S: RNA5S1, RNA5S2, RNA5S3, RNA5S4, RNA5S5, RNA5S6, RNA5S7, RNA5S8, RNA5S9, RNA5S10, RNA5S11, RNA5S12, RNA5S13S, RNA5S
- Mt: MT-RNR1, MT-TV(공동작업), MT-RNR2
「 」를 참조해 주세요.
레퍼런스
- ^ a b Berk, Arnold; Baltimore, David; Lodish, Harvey; Darnell, James; Matsudaira, Paul; Zipursky, S. Lawrence (1996-01-31). Molekulare Zellbiologie. Berlin, Boston: DE GRUYTER. doi:10.1515/9783110810578. ISBN 9783110810578.
- ^ Lodish, Harvey; Berk, Arnold; Zipursky, S. Lawrence; Matsudaira, Paul; Baltimore, David; Darnell, James (2000). "The Three Roles of RNA in Protein Synthesis". Molecular Cell Biology. 4th Edition.
- ^ Urlaub H, Kruft V, Bischof O, Müller EC, Wittmann-Liebold B (September 1995). "Protein-rRNA binding features and their structural and functional implications in ribosomes as determined by cross-linking studies". The EMBO Journal. 14 (18): 4578–88. doi:10.1002/j.1460-2075.1995.tb00137.x. PMC 394550. PMID 7556101.
- ^ Ferreira-Cerca S, Pöll G, Gleizes PE, Tschochner H, Milkereit P (October 2005). "Roles of eukaryotic ribosomal proteins in maturation and transport of pre-18S rRNA and ribosome function". Molecular Cell. 20 (2): 263–75. doi:10.1016/j.molcel.2005.09.005. PMID 16246728.
- ^ Szymański M, Barciszewska MZ, Erdmann VA, Barciszewski J (May 2003). "5 S rRNA: structure and interactions". The Biochemical Journal. 371 (Pt 3): 641–51. doi:10.1042/bj20020872. PMC 1223345. PMID 12564956.
- ^ Henras AK, Plisson-Chastang C, O'Donohue MF, Chakraborty A, Gleizes PE (2015-03-01). "An overview of pre-ribosomal RNA processing in eukaryotes". Wiley Interdisciplinary Reviews: RNA. 6 (2): 225–42. doi:10.1002/wrna.1269. PMC 4361047. PMID 25346433.
- ^ Penev PI, Fakhretaha-Aval S, Patel VJ, Cannone JJ, Gutell RR, Petrov AS, Williams LD, Glass JB (August 2020). "Supersized ribosomal RNA expansion segments in Asgard archaea". Genome Biology and Evolution. 12 (10): 1694–1710. doi:10.1093/gbe/evaa170. PMC 7594248. PMID 32785681.
- ^ Luehrsen, KR.; Nicholson, DE; Eubanks, DC; Fox, GE (May 1981). "An archaebacterial 5S rRNA contains a long insertion sequence". Nature. 293 (5835): 755–756. Bibcode:1981Natur.293..755L. doi:10.1038/293755a0. PMID 6169998. S2CID 4341755.
- ^ Tirumalai, MR; Kaelber, JT; Park, DR; Tran, Q; Fox, GE (31 August 2020). "Cryo‐electron microscopy visualization of a large insertion in the 5S ribosomal RNA of the extremely halophilic archaeon Halococcus morrhuae". FEBS Open Bio. 10 (10): 1938–1946. doi:10.1002/2211-5463.12962. PMC 7530397. PMID 32865340.
- ^ Woese CR, Fox GE (November 1977). "Phylogenetic structure of the prokaryotic domain: the primary kingdoms". Proc. Natl. Acad. Sci. USA. 74 (11): 5088–5090. Bibcode:1977PNAS...74.5088W. doi:10.1073/pnas.74.11.5088. PMC 432104. PMID 270744.
- ^ Lagesen K, Hallin P, Rødland EA, Staerfeldt HH, Rognes T, Ussery DW (2007-05-01). "RNAmmer: consistent and rapid annotation of ribosomal RNA genes". Nucleic Acids Research. 35 (9): 3100–8. doi:10.1093/nar/gkm160. PMC 1888812. PMID 17452365.
- ^ Chun J, Lee JH, Jung Y, Kim M, Kim S, Kim BK, Lim YW (October 2007). "EzTaxon: a web-based tool for the identification of prokaryotes based on 16S ribosomal RNA gene sequences". International Journal of Systematic and Evolutionary Microbiology. 57 (Pt 10): 2259–61. doi:10.1099/ijs.0.64915-0. PMID 17911292.
- ^ a b c Tirumalai MR, Rivas M, Tran Q, Fox GE (November 2021). "The Peptidyl Transferase Center: a Window to the Past". Microbiol Mol Biol Rev. 85 (4): e0010421. doi:10.1128/MMBR.00104-21. PMC 8579967. PMID 34756086.
- ^ Ghosh, Arnab; Komar, Anton A (2 January 2015). "Eukaryote-specific extensions in ribosomal proteins of the small subunit: Structure and function". Translation. 3 (1): e999576. doi:10.1080/21690731.2014.999576. PMC 4682806. PMID 26779416.
- ^ Lodmell JS, Dahlberg AE (August 1997). "A conformational switch in Escherichia coli 16S ribosomal RNA during decoding of messenger RNA". Science. 277 (5330): 1262–7. doi:10.1126/science.277.5330.1262. PMID 9271564.



